Red booleana
Una Red Booleana se compone de un conjunto de variables booleanas cuyo estado está determinado por otras variables en la red. Son un caso particular de red dinámica discreta, donde el tiempo y los estados son discretos, es decir, tienen una biyección sobre una serie entera. Los autómatas celulares booleanos y elementales son casos particulares de las redes booleanas, donde se determina el estado de una variable por sus vecinos espaciales.
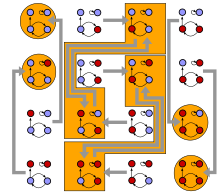
En una red booleana aleatoria las conexiones son decididas al azar y las salidas de los nodos están determinadas por funciones lógicas generadas al azar.
Red booleana aleatoria clásica editar
Las redes booleanas aleatorias (RBN) se conocen como redes NK o redes de Kauffman. Las redes booleanas aleatorias fueron propuestas como modelos de las redes de regulación génica por Stuart Alan Kauffman en 1969 (Kauffman, 1969, 1993).
Una RBN es un sistema de N nodos de estado binario (que representan los genes) con K entradas en cada nodo que representa los mecanismos de regulación. Los dos estados (encendido / apagado) representan, respectivamente, la condición que un gen se activa o inactiva. La variable K suele mantenerse constante, pero también puede ser variada a través de todos los genes, transformándola en un conjunto de enteros en lugar de un único entero. En el caso más simple, a cada gen se le asigna al azar K entradas regulatorias de entre los N genes y una de las posibles funciones booleanas de K entradas. Esto da una muestra aleatoria de los posibles conjuntos de las redes NK. El estado de una red en cualquier punto en el tiempo está dado por los estados actuales de todos los N genes. Así, el espacio de estados de cualquier red, es 2N.
La simulación de las RBN se realiza en pasos de tiempo discretos. El estado de un nodo en el tiempo t +1 se calcula mediante la aplicación de la función booleana asociada con el nodo al estado de sus nodos de entrada en el momento t. El comportamiento de RBN específicas y clases generalizadas de ellas ha sido objeto de gran parte de la investigación de Kauffman (y de otros).
Esquemas de Actualización editar
Las RBN clásicas (CRBN) utilizan un esquema de actualización sincrónico y una crítica de las CRBN como modelos de las redes de regulación génica es que los genes no cambian todos sus estados en el mismo momento. Harvey y Bossomaier presentaron esta crítica y definieron RBN asincrónicas (ARBN) en donde se selecciona y actualiza un nodo al azar en cada instante de tiempo (Harvey y Bossomaier, 1997). Ya que un nodo se actualiza al azar, las ARBN no son deterministas y no tienen los atractores de ciclo que se encuentran en CRBN (Gershenson, 2004).
Las RBN asincrónicas deterministas (DARBN) fueron presentados por Gershenson como una manera de tener RBN que no tienen la actualización sincrónica, pero aún son deterministas. En DARBN cada nodo tiene dos parámetros generados al azar Pi and Qi (Pi, Qi ∈ ℕ, Pi > Qi). Estos parámetros permanecen fijos. Un nodo i se actualizará cuando t ≡ Qi (mod Pi) donde t es el instante de tiempo. Si más de un nodo debe ser actualizado en un instante de tiempo se actualizan los nodos en un orden predefinido, por ejemplo, de menor a mayor i. Otra forma de hacer esto es actualizar de forma sincrónica todos los nodos que cumplen la condición de actualización. Este último esquema se llama RBN semi-sincrónico determinista o asincrónico generalizado determinista (DGARBN) (Gershenson, 2004).
Las RBN en donde uno o más nodos son seleccionados para la actualización en cada instante de tiempo y los nodos seleccionados se actualizan de forma sincrónica se llaman RBN asincrónicas generalizadas (GARBN). Las GARBN son semi-sincrónicas, pero no deterministas (Gershenson, 2002).
Atractores editar
Una red booleana tiene 2N posibles estados. Tarde o temprano va a llegar a un estado visitado anteriormente y, por lo tanto, ya que la dinámica es determinista, cae en un atractor. Si el atractor es un único estado, se denomina punto atractor, y si el atractor se compone de más de un estado, se denomina un ciclo atractor. El conjunto de estados que conducen a un atractor se llama cuenca del atractor. Los estados sin predecesores se llaman estados jardín del Edén y la dinámica de la red fluye desde estos estados hacia los atractores. El tiempo que se tarda en llegar a un atractor es llamado tiempo transiente. (Gershenson, 2004)
Atractores de distintas redes booleanas bajo actualización paralela editar
| Características de la red | Características de los puntos fijos | Características de los ciclos límite | |
|---|---|---|---|
| General (sin restricciones) | Determinar si existe es NP-completo (Zhao,). Encontrar uno es NP-difícil (Akutsu, 1998) | Encontrarlos es NP-completo | |
| Sin circuitos | Único. Se puede encontrar en tiempo polinomial (F. Robert,) | ||
| Sin circuitos negativos | Existen (Aracena,) | ||
| K=N | (Rubin,1954) | ||
| Monótona | Encontrar el número es NP-completo (Zhao,) | (Teorema de Sperner) | |
| Sin circuitos de largo (grafo por niveles) | , p ∈ ℕ (Goles, Salinas, 2007) | ||
| Sin circuitos de largo y ausencia de ciclos no monótonos | No existen (Goles, Salinas, 2007) | ||
| Regulatoria | número (Aracena, 2008) | ||
| Caterpillar monótona | (Aracena, 2004) | ||
| Fuertemente conectada y todos sus circuitos son positivos | Existen al menos dos (Aracena, 2008) |
Notación: k: Largo de los circuitos. K: Número de arcos que llegan a cada nodo. L: Largo de los ciclos. n, N: Número de nodos. τ(G): Número mínimo de vértices del conjunto feedback vertex set. τ : Lo anterior pero para ciclos positivos.
Topologías editar
- Homogénea
- Normales
- Red libre de escala (Aldana, 2003)
Aplicaciones editar
Referencias editar
- Aldana, M. (2003). *Boolean dynamics of networks with scale-free topology. Physica D 185:45–66
- Aldana , M., Coppersmith, S., and Kadanoff, L. P. (2003). Boolean dynamics with random couplings. In Kaplan, E., Marsden, J. E., and Sreenivasan, K. R., editors, Perspectives and Problems in Nonlinear Science. A Celebratory Volume in Honor of Lawrence Sirovich. Springer Applied Mathematical Sciences Series.
- Kauffman, S. A. (1969). Metabolic stability and epigenesis in randomly constructed genetic nets. Journal of Theoretical Biology, 22:437-467.
- Kauffman, S. A. (1993). Origins of Order: Self-Organization and Selection in Evolution. Oxford University Press. Technical monograph. ISBN 0-19-507951-5
- Gershenson, C. (2002). *Classification of random Boolean networks. In Standish, R. K., Bedau, M. A., and Abbass, H. A., editors, Artificial Life VIII:Proceedings of the Eight International Conference on Artificial Life, pages 1-8. MIT Press.
- Gershenson, C (2004). *Introduction to Random Boolean Networks Carlos Gershenson, editors M. Bedau and P. Husbands and T. Hutton and S. Kumar and H. Suzuki, “Workshop and Tutorial Proceedings, Ninth International Conference on the Simulation and Synthesis of Living Systems {(ALife} {IX)}”, pages 160–173.
- Harvey, I. and Bossomaier, T. (1997). Time out of joint: Attractors in asynchronous random Boolean networks. In Husbands, P. and Harvey, I., editors, Proceedings of the Fourth European Conference on Artificial Life (ECAL97), pages 67-75. MIT Press.
- Wuensche, A. (1998). *Discrete dynamical networks and their attractor basins. In Standish, R., Henry, B., Watt, S., Marks, R., Stocker, R., Green, D., Keen, S., and Bossomaier, T., editors, Complex Systems'98, University of New South Wales, Sydney, Australia.