Citoesqueleto procariota
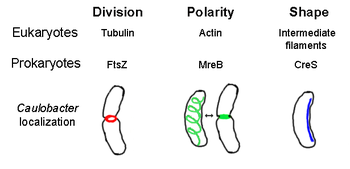
El citoesqueleto procariota es el nombre colectivo para todos los filamentos estructurales en procariontes. Se creía que las células de procariotas no lo poseían citoesqueletos, pero avances en tecnología de visualización y determinación de estructura, llevaron al descubrimiento de filamentos en estas células en 1990.[2] No sólo tienen equivalentes para proteínas en el citoesqueleto en eucariontes que fueron encontrados en procariontes,[3] Los elementos del citoesqueleto juegan funciones esenciales en la división de célula, protección, determinación de forma, y determinación de la polaridad en varios procariotas.[4][5]
FtsZ
editarFtsZ, el primer citoesqueleto identificado, el cual forma una estructura filamentosa de anillo localizada en medio de la célula conocida como anillo Z que se contrae durante la división de célula, similar a la actina-miosina anillo contráctil en eucariontes.[2] El Z-anillo es una estructura altamente dinámica que consta de fardos numerosos de protofilamentos que extienden y encogen, a pesar de que el mecanismo detrás de anillo Z y el número de protofilamentos implicó son unicelulares.[1] FtsZ Actúa como una proteína de organización y se requiere para la división de célula. Es el primer componente del tabique durante citoquinesis, y quien recluta todo para la célula durante la división en el sitio de división.[6]
A pesar de esta semejanza funcional para la actinia, FtsZ es homólogo a la tubulina eucariota. Aunque la comparación de las estructuras primarias de FtsZ y tubulina revelan una relación débil, sus estructuras tridimensionales son notablemente similares. Además, como tubulina monomérica, FtsZ está ligada a GTP y se polimeriza con otros monómeros FtsZ con la hidrólisis de GTP en un mecanismo similar a la dimerización de tubulina. [7] puesto que la FtsZ es esencial para la división celular en bacterias, esta proteína es un objetivo para el diseño de nuevos antibióticos. [8] Actualmente existen varios modelos y mecanismos que regulan la formación del anillo Z. Uno del modelo de formación del anillo Z, fue propuesto por Rashid, Aijaz y canta, Perminder. Modelo de gradiente de la concentración crítica de la división celular bacteriana. De naturaleza precedentes < http://hdl.handle.net/10101/npre.2010.4659.1 > (2010). Según este modelo los reguladores positivos y negativos de FtsZ Asamblea forman un gradiente dentro de la célula bacteriana. El gradiente es tal que la concentración crítica de FtsZ requerida para el montaje de FtsZ es mínima en posición medial de la célula, por lo tanto, el anillo Z y finalmente el divisoma complejo están dispuestos en medio de la célula.
MreB
editarMreB es una proteína bacteriana creída que ser análoga a la actina eucariota. MreB y actina tienen un partido débil estructura primaria, pero son muy similares en términos de polimerización tridimensional de estructura y filamento.
Casi todas las bacterias no esféricas dependen de MreB para determinar su forma. MreB reúne en una red helicoidal de estructuras filamentosas justo debajo de la membrana citoplasmática, cubriendo toda la longitud de la célula. [9] MreB determina la forma de la célula mediando la posición y la actividad de las enzimas que sintetizan peptidoglucano y actuando como un filamento rígido debajo de la membrana celular que ejerce presión hacia fuera para esculpir y reforzar la célula. [1] MreB se condensa desde su red helicoidal normal y forma un anillo apretado en el septo en Caulobacter crescentus justo antes de la división celular, un mecanismo que se cree para ayudar a localizar su tabique en el centro. [10] MreB también es importante para la determinación de la polaridad en bacterias polares, es responsable de la correcta posición de por lo menos cuatro diferentes proteínas polares en C. crescentus. [10]
Crescentina
editarCrescentina (codificada por el gen creS) es un análogo de filamentos intermedios eucariotas (IFs). A diferencia de las otras relaciones análogas discutidas aquí, la crescentina tiene una homología primaria grande con proteínas IF, además de similitud tridimensional - la secuencia de creS tiene un porcentaje de similitud del 25% y 40% con la citoqueratina 19 y una similitud de fósforo y 40% 24% de la identidad nuclear lamin A. Además, los filamentos de crescentina tienen aproximadamente 10 nanómetros de diámetro y por lo tanto caen dentro de la gama de diámetro para las IFs eucariotas (8-15 nm). [11] La crescentina forma un filamento continuo de polo a polo junto con el lado interno cóncavo de la bacteria en forma de medialuna Caulobacter crescentus. Tanto MreB como crescentina son necesarios para que C. crescentus existan en su forma característica; se cree que moldes de MreB de la célula en forma de barra y crescentina curvas de esta forma en una media luna. [1].
Véase también
editar- División de célula
- Cytokinesis
- Citoesqueleto
- Prokaryotes
- Filamento de proteína
Referencias
editar- ↑ a b Gitai, Z. (2005). «The New Bacterial Cell Biology: Moving Parts and Subcellular Architecture». Cell 120 (5): 577-586. PMID 15766522. doi:10.1016/j.cell.2005.02.026.
- ↑ a b Bi, E.; Lutkenhaus, J. (1991). «FtsZ ring structure associated with division in Escherichia coli». Nature 354 (6349): 161-164. PMID 1944597. doi:10.1038/354161a0.
- ↑ Wickstead B, Gull K (2011). «The evolution of the cytoskeleton». The Journal of Cell Biology 194 (4): 513-25. PMC 3160578. PMID 21859859. doi:10.1083/jcb.201102065.
- ↑ Shih YL, Rothfield L (2006). «The bacterial cytoskeleton». Microbiol. Mol. Biol. Rev. 70 (3): 729-54. PMC 1594594. PMID 16959967. doi:10.1128/MMBR.00017-06.
- ↑ Michie KA, Löwe J (2006). «Dynamic filaments of the bacterial cytoskeleton». Annu. Rev. Biochem. 75: 467-92. PMID 16756499. doi:10.1146/annurev.biochem.75.103004.142452. Archivado desde el original el 26 de marzo de 2009. Consultado el 2 de mayo de 2015.
- ↑ Graumann, P.L. (2004). «Cytoskeletal elements in bacteria». Current Opinion in Microbiology 7 (6): 565-571. PMID 17506674. doi:10.1016/j.mib.2004.10.010.