Cromatina
La cromatina[1] es la forma en la que se hace observable el material genético en el núcleo celular. Es la sustancia de los cromosomas y corresponde a la asociación de ADN, ARN y proteínas que se encuentran en el núcleo en interfase de las células. Las proteínas son de dos tipos: las histonas y las proteínas no histónicas.
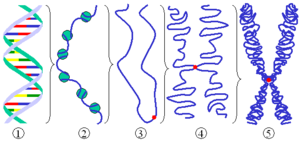
Las unidades básicas de la cromatina son los nucleosomas. Estos se encuentran formados por aproximadamente 146 pares de bases de longitud (el número depende del organismo), asociados a un complejo específico de ocho histonas nucleosómicas (octámero de histonas). Cada partícula tiene una forma de disco, con un diámetro de 11 nanómetros (nm) y contiene dos copias de cada una de las cuatro histonas H3, H4, H2A y H2B. Este octámero forma un núcleo proteico, alrededor del cual se enrolla la hélice de ADN (de aproximadamente 1,8 vueltas). Entre cada una de las asociaciones de ADN e histonas existe un ADN libre llamado ADN espaciador, de longitud variable entre 0 y 80 pares de nucleótidos que garantiza flexibilidad a la fibra de cromatina. Este tipo de organización, permite un primer paso de compactación del material genético, y da lugar a una estructura parecida a un "collar de perlas".
Posteriormente, un segundo nivel de organización de orden superior lo constituye la "fibra de 30 nm", compuesta por grupos de nucleosomas empaquetados unos sobre otros adoptando disposiciones regulares gracias a la acción de la histona H1.
Finalmente, continúa el incremento del empaquetamiento del ADN hasta obtener los cromosomas que se observan en la metafase, este es el máximo nivel de condensación del ADN.
Historia
editarEn 1879 Walther Flemming fue el primero en describir la cromatina; le otorgó este nombre debido a su afinidad por los colorantes. Sugirió que podía ser idéntica a la nucleína.[2]
En 1869 Miescher había aislado una sustancia molecular fosforada a la que llamó nucleína. En 1889 Altman la caracterizaría como ácido orgánico y la llamaría ácido nucleico, hoy ADN.[3]
Las histonas fueron descubiertas poco después, en 1884, por Albrecht Kossel. Pocos progresos se realizaron en la determinación de la estructura de la cromatina hasta la década de 1970, cuando se pudieron hacer las primeras observaciones de fibras de cromatina por microscopía electrónica, revelando la existencia del nucleosoma, la unidad de base de la cromatina, cuya estructura detallada fue finalmente resuelta por cristalografía de rayos X en 1997.[4]
Tipos de cromatina
editarLa cromatina se puede encontrar en dos formas: Heterocromatina y Eucromatina.
Eucromatina
editarEs una forma de la cromatina que está ligeramente compactada, que muestra una gran concentración de genes, es la forma activa. Está formada por una fibra de un diámetro que corresponde al del nucleosoma, que es un segmento de ADN bicatenario enrollado alrededor de homodímeros de las histonas H2A, H2B, H3, y H4. En la eucromatina inactiva, esta fibra se enrolla sobre sí misma gracias a las histonas H1 para formar el solenoide. La interacción con otras proteínas no histonas (topoisomerasa II, proteínas de andamiaje, lamininas, …) provoca mayores grados de organización. En cuanto a la heterocromatina, la fibra que la constituye se encuentra más condensada y a menudo aparece formada por agregados. Su formación require numerosas proteínas adicionales, que incluyen las proteínas HP1 (Heterochromatin Protein 1 o proteína de la heterocromatina1).
Heterocromatina
editarEs una forma inactiva condensada localizada sobre todo en la periferia del núcleo celular, que se tiñe fuertemente con las coloraciones de histología. En 1928, Emil Heitz, basándose en observaciones histológicas, definió la heterocromatina como los segmentos cromosómicos que aparecían muy condensados y oscuros en el núcleo en interfase. De hecho, la cromatina está formada de una maraña de fibras cuyo diámetro no solo varía durante el ciclo celular sino que también depende de la región del cromosoma observada.
La heterocromatina puede ser de dos tipos diferentes, la riqueza en ADN satélite determina tanto la naturaleza permanente o reversible de la heterocromatina, como su polimorfismo y propiedades de tinción.:
- la constitutiva, idéntica para todas las células del organismo y que carece de información genética, incluye a los telómeros y centrómeros del cromosoma que no expresan su ADN. La heterocromatina constitutiva contiene un tipo particular de ADN denominado ADN satélite, formado por gran número de secuencias cortas repetidas en tándem. Los tipos principales de este ADN son el ADN satélite alfa, y los ADN satélite I, II y III. Estas secuencias de ADN satélite son capaces de plegarse sobre sí mismas y pueden tener un papel importante en la formación de la estructura altamente compacta de la heterocromatina constitutiva. La heterocromatina constitutiva es estable y conserva sus propiedades heterocromáticas durante todas las etapas del desarrollo y en todos los tejidos. La heterocromatina constitutiva es altamente polimórfica, probablemente debido a la inestabilidad del ADN satélite. Este polimorfismos puede afectar, no solamente a su tamaño sino también a la localización de la heterocromatina, y aparentemente no tiene un efecto fenotípico. La heterocromatina constitutiva se encuentra fuertemente teñida en la técnica de bandas C, lo que es el resultado de una renaturalización muy rápida del ADN satélite tras la desnaturalización.
- la facultativa, diferente en los distintos tipos celulares, contiene información sobre todos aquellos genes que no se expresan o que pueden expresarse en algún momento. Incluye al ADN satélite y al corpúsculo de Barr. La heterocromatina facultativa se caracteriza por la presencia de secuencias repetidas tipo LINE. Estas secuencias, dispersas a lo largo del genoma, podrían promover la propagación de una estructura de cromatina condensada. La heterocromatina facultativa es reversible, su estado heterocromático depende de la etapa del desarrollo y del tipo celular. Dos ejemplos de este tipo de heterocromatina son el cromosoma X inactivo (cuerpo de Barr) de las células somáticas femeninas y la vesícula sexual inactiva en la etapa del paquiteno de las meiosis masculinas. La heterocromatina facultativa no es particularmente rica en ADN satélite, y por ello, no es polimórfica. La heterocromatina facultativa no se encuentra nunca teñida en la técnica de bandas C.
Se ha visto que en la formación de heterocromatina frecuentemente participa el fenómeno de ARN interferente. Por ejemplo, en Schizosaccharomyces pombe, la heterocromatina se forma en el centrómero, telómeros y en el loci mating-type.[5] La formación de la heterocromatina en el centrómero depende del mecanismo de ARN interferente (ARNi). ARN doble cadena complementarios son producidos de secuencias repetidas localizadas en el centrómero, que inducen ARNi y seguidamente metilación de la lisina 9 histona 3 y enlazamiento de Swi6 (proteína estructural de la heterocromatina, la cual es homóloga a HP1 en mamíferos).[6]
Propiedades de la heterocromatina
editarA pesar de las diferencias descritas anteriormente, la heterocromatina constitutiva y la heterocromatina facultativa tienen propiedades muy similares.
1. La heterocromatina está condensada. Este es, de hecho, lo que define la heterocromatina, y por ello es aplicable tanto a la heterocromatina constitutiva como a la facultativa. Esta elevada condensación la hace fuertemente cromofílica e inaccesible a la DNAsa I y, en general, a otras enzimas de restricción.
2. El ADN de la heterocromatina se replica más tarde.
La incorporación de varios análogos de nucleótidos muestra que el ADN de ambos tipos de heterocromatina se replica tarde. Esto es el resultado, por un lado, de su elevado grado de condensación, que evita que la maquinaria replicativa acceda fácilmente al ADN y, por otro lado, de su localización en un dominio nuclear periférico pobre en elementos activos.
3. El ADN de la heterocromatina se encuentra metilado.
- El ADN de la heterocromatina constitutiva se encuentra altamente metilado en las citosinas. Por ello, un anticuerpo anti-5-metil citosina marca fuertemente todas las regiones de este tipo de heterocromatina.
- Por lo que se refiere a la heterocromatina facultativa, la metilación de su ADN es menor, aunque los análisis mediante enzimas de restricción sensibles a metilación revelan una importante metilación de los islotes CpG, específicamente localizados en las regiones que controlan la expresión de los genes.
4. En la heterocromatina las histonas se encuentran hipoacetiladas. Las histonas puede sufrir una serie de modificaciones post-traduccionales en sus extremos N-terminales que pueden afectar a la propia actividad genética de la cromatina.
- La hipoacetilación de las colas N-terminales de las histonas, principalmente en las lisinas, están asociadas con la cromatina inactiva. Por el contrario, las histonas hiperacetiladas son características de la cromatina activa.
- La acetilación/desacetilación de histonas es un mecanismos absolutamente esencial para el control de la expresión génica. Existen numerosos factores de transcripción que presentan una actividad acetiltransferasa de histonas (HAT, Histone Acetyl Transferase) o desacetilasa de histonas (HDAc o Histone De-Acetylase).
5. Las histonas de la heterocromatina se encuentran metiladas en la lisina 9. La metilación de la lisina 9 de la histona H3 (H3-K9) parece que está muy relacionada con el proceso de heterocromatinización del genoma, tanto en la formación de heterocromatina constitutiva como facultativa.
6. La heterocromatina es transcripcionalmente inactiva.
- A diferencia de lo que ocurre en Drosophila, la heterocromatina constitutiva humana no contiene genes y la incorporación de uridina tritiada en los cultivos celulares no producen ningún tipo de marcaje a este nivel.
- La heterocromatina facultativa es relativamente pobre en genes, y éstos generalmente no se transcriben en el estado de heterocromatina.
7. La heterocromatina no participa en la recombinación genética.
- De modo general se acepta que la heterocromatina constitutiva no participa en la recombinación genética. La no existencia de un emparejamiento preliminar de las regiones heterocromatínicas homólogas se podría deber al polimorfismo característico de estas regiones que lo dificultarían, aunque no lo harían imposible. La heterocromatina constitutiva también actúa reprimiendo la recombinación en las regiones de eucromatina adyacentes.•Por lo que respecta a la heterocromatina facultativa, tampoco participa en la recombinación meiótica cuando se encuentra en su forma inactiva.
Funciones de la heterocromatina
editarDurante mucho tiempo el papel concreto de la heterocromatina ha sido un misterio, ya que su polimorfismo no parecía tener ningún efecto funcional o fenotípico.
- . Papel de la heterocromatina en la organización de los dominios nucleares.
- La heterocromatina y la eucromatina ocupan dominios nucleares distintos. La heterocromatina se localiza generalmente en la periferia del núcleo anclada a la envoltura nuclear. Por el contrario, la cromatina activa se localiza en una posición central.
- La localización preferencial de la heterocromatina contra la envoltura nuclear puede deberse a la interacción de la proteína HP1 con el receptor de la lamina B, componente de la lámina nuclear, una estructura proteica que es adyacente a la envoltura nuclear por su cara interna. •La localización periférica de la heterocromatina concentra los elementos activos en la porción central del núcleo, permitiendo que eucromatina activa se replique y transcriba con una eficiencia máxima.
- . Papel de la heterocromatina en la función del centrómero.
En la mayor parte de eucariotas, los centrómeros se encuentran rodeados de una considerable masa de heterocromatina. Se ha sugerido que la heterocromatina centromérica sería necesaria para la cohesión de las cromátidas hermanas y que permitiría la disyunción normal de los cromosomas mitóticos.
- En la levadura Schizosaccharomyces pombe, el homólogo Swi6 de la proteína HP1 es absolutamente esencial para la cohesión eficiente de las cromátidas hermanas durante la división celular.
- Los experimentos en los cuales se ha realizado la deleción del ADN satellite muestran que una gran región de repeticiones de este tipo de ADN es indispensable para el funcionamiento correcto del centrómero.
Se supone que la heterocromatina centromérica podría, de facto, crear un compartimento mediante el incremento de la concentración local de la variante centromérica de las histonas, CENP-A, y mediante la promoción de la incorporación de la CENP-A en lugar de la histona H3 durante la replicación.
- . Papel de la heterocromatina en la represión génica (regulación epigenética)
La expresión génica puede estar controlada a dos niveles:
- Primero, a nivel local o control transcripcional, gracias a la formación de complejos locales de transcripción. Este nivel involucra secuencias de ADN relativamente pequeñas unidas a genes.
- A nivel más global, en cuyo caso se dice que hay un control de la transcriptabilidad. Este control involucra a secuencias más largas que representan un gran dominio de cromatina, que puede estar en estado activo o inactivo. En este caso es la heterocromatina la que parece estar involucrada. Los genes que generalmente se encuentran en la eucromatina pueden, por tanto, ser silenciados cuando se encuentran cercanos a un dominio de heterocromatina.
Mecanismo de inactivación en cis:
Los reordenamientos cromosómicos pueden provocar que una región eucromática se yuxtaponga a una región heterocromática. En el momento en el que el reordenamiento elimina ciertas barreras que protegen la eucromatina la estructura heterocromática es capaz de propagarse en cis a la eucromatina adyacente, inactivando los genes que se encuentran en ella. Este es el mecanismo observado en la variegación por efecto de posición (PEV) en Drosophila y en la inactivación de ciertos transgenes en ratón.
Mecanismo de inactivación en trans:
Durante la diferenciación celular, ciertos genes activos pueden transponerse a un dominio nuclear heterocromático haciendo que se inactiven. Este mecanismo es el que se ha propuesto como explicación para la co-localización en los núcleos de linfocitos de la proteína IKAROS con la heterocromatina centromérica y de los genes cuya expresión controla.
- Eucromatina, está diseminada por el resto del núcleo (menor condensación), se tiñe débilmente con la coloraciones (su mayor tinción ocurre en la mitosis y no es visible con el microscopio de luz). Representa la forma activa de la cromatina en la que se está transcribiendo el material genético de las moléculas de ADN a moléculas de ARNm, por lo que es aquí donde se encuentran la mayoría de los genes activos.
Rol de la cromatina en la expresión genética
editarLa cromatina es una estructura dinámica que adapta su estado de compactación y empaquetamiento para optimizar los procesos de replicación, transcripción y reparación del ADN, juega un rol regulatorio fundamental en la expresión génica. Los distintos estados de compactación pueden asociarse (aunque no unívocamente) al grado de transcripción que exhiben los genes que se encuentran en esas zonas. La cromatina es, en principio, fuertemente represiva para la transcripción, ya que la asociación del ADN con las distintas proteínas dificulta la procesión de las distintas ARN polimerasas. Por lo tanto, existe una variada cantidad de máquinas remodeladoras de la cromatina y modificadoras de histonas.
Existe actualmente lo que se conoce como "código de histonas". Las distintas histonas pueden sufrir modificaciones post-traduccionales, como ser la metilación, acetilación, fosforilación, generalmente dada en residuos lisina o arginina. La acetilación está asociada con activación de la trascripción, ya que al acetilarse una lisina, disminuye la carga positiva global de la histona por lo cual tiene una menor afinidad por el ADN (que está cargado negativamente). En consecuencia, el ADN se encuentra unido menos fuertemente lo que permite el acceso de la maquinaria transcripcional. Por el contrario, la metilación está asociada con la represión transcripcional y una unión ADN-histona más fuerte (si bien no siempre esto se cumple). Por ejemplo, en la levadura S. pombe, la metilación en el aminoácido (residuo 9 ) de lisina de la histona H3, está asociado con represión de la transcripción en la heterocromatina, mientras que la metilación en el (residuo 4) de lisina, promueve la expresión de genes.[6]
Las enzimas que llevan a cabo las funciones de modificaciones de histonas son las acetilasas y desacetilasas de histonas, y las metilasas y desmetilasas de histonas, que forman distintas familias cuyos integrantes se encargan de modificar un residuo en particular de la larga cola de las histonas.
Además de las modificaciones de las histonas, existen también maquinarias remodeladoras de la cromatina, como por ejemplo SAGA, que se encargan de reposicionar nucleosomas, ya sea desplazándolos, rotándolos, o incluso desensamblándolos parcialmente, retirando algunas de las histonas constituyentes del nucleosoma y luego volviéndolos a colocar. En general las maquinarias remodeladoras de la cromatina son esenciales para el proceso de transcripción en eucariotas, ya que permiten el acceso y procesividad de las polimerasas.
Otra forma de marcación de la cromatina como "inactiva" puede darse a nivel de la Metilación del ADN, en citosinas que pertenezcan a dinucleótidos CpG. En general la metilación del ADN y de la cromatina son procesos sinérgicos, ya que, por ejemplo, al metilarse el ADN, existen enzimas metiladoras de histonas que pueden reconocer citosinas metiladas, y metilan histonas próximas. Del mismo modo, enzimas que metilan el ADN pueden reconocer histonas metiladas, y así seguir con la metilación a nivel de ADN.
Todas estas modificaciones forman parte de la familia de las modificaciones epigenéticas.
Cromatina y mutaciones
editarLa carga de mutaciones es mayor en aquellas zonas que presentan cromatina reprimida y en regiones donde la replicación es tardía, lo cual se ha observado también en cáncer humano.
Métodos de captura de la conformación de los cromosomas
editarLos métodos de captura de la conformación de los cromosomas (normalmente llamados también tecnología 3C) son métodos utilizados en biología molecular para determinar la organización espacial de la cromatina en la célula. Estos métodos cuantifican el número de interacciones de loci genómicos que están cerca en le estructura 3D, pero pueden encontrase lejos respecto a la secuencia nucleotídica.Se han desarrollado muchos métodos para estudiar estos contactos genómicos y todos comparten unos pasos iniciales:[7] 1. Entrecruzamiento del DNA con formaldehído. 2. Corte del genoma mediante endonucleasas 3. Ligación de los fragmentos de DNA al azar. la ligación de los fragmentos entrecruzados está favorecida frente a los fragmentos sueltos debido a la cercanía de los fragmentos entrecruzados. 4. Posteriormente, se analizan los fragmentos obtenidos mediante diferentes técnicas de PCR.
Dip-c:[8] método de estudio de la estructura de la cromatina basado en una modificación de la técnica anterior de conformación de la cromatina, Hi-C, combinado con una amplificación de todo el genoma con alta cobertura por múltiple amplificación de end-tagging llamado META. En concreto, se modifica el paso de ligación con biotina del método Hi-C. Este tipo de técnicas requiere un análisis bioinformático posterior. En Dip-C, se establece un algoritmo con base en la hipótesis del vecindario. Ésta postula que dos haplotipo homólogos deben tener diferentes patrones de contacto y que, por tanto, los haplotipos desconocidos de un cromosoma deben contactar en una región cromosómica cercana o vecina. Los autores definen el vecindario como una superelipse con un exponente de 0.5 y un radio de 10 Mb. Este método, a diferencia de los anteriores (3C, 4C, 5C y Hi-C), permite analizar la estructura de la cromatina de células diploides basándose en polimorfismos de un único nucleótido o SNPs. De este modo, se consigue establecer qué haplotipo celular está implicado en cada contacto cromosómico. El estudio llevado con este método confirma que detecta más contactos entre los cromosomas y un menor número de falsos positivos.
Véase también
editarReferencias
editar- ↑ OMS,OPS,BIREME (ed.). «Cromatina». Descriptores en Ciencias de la Salud. Biblioteca Virtual en Salud.
- ↑ Olins, Donald E.; Olins, Ada L. (Octubre de 2003). «Chromatin history: our view from the bridge». Nature Reviews. Molecular Cell Biology 4 (10): 809-814. ISSN 1471-0072. PMID 14570061. doi:10.1038/nrm1225. Consultado el 8 de marzo de 2023.
- ↑ Deichmann U. (2015). «Chromatin: Its history, current research, and the seminal researchers and their philosophy». Perspectives in Biology and Medicine (ResearchGate) 58 (2): 143-164. Consultado el 16 de junio de 2024.
- ↑ Luger, K.; Mäder, A. W.; Richmond, R. K.; Sargent, D. F.; Richmond, T. J. (18 de septiembre de 1997). «Crystal structure of the nucleosome core particle at 2.8 A resolution». Nature 389 (6648): 251-260. ISSN 0028-0836. PMID 9305837. doi:10.1038/38444. Consultado el 8 de marzo de 2023.
- ↑ Lippman, Zachary; Martienssen, Rob (16 de septiembre de 2004). «The role of RNA interference in heterochromatic silencing». Nature 431 (7006): 364-370. ISSN 1476-4687. PMID 15372044. doi:10.1038/nature02875. Consultado el 8 de marzo de 2023.
- ↑ a b Volpe, Thomas A.; Kidner, Catherine; Hall, Ira M.; Teng, Grace; Grewal, Shiv I. S.; Martienssen, Robert A. (13 de septiembre de 2002). «Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi». Science (New York, N.Y.) 297 (5588): 1833-1837. ISSN 1095-9203. PMID 12193640. doi:10.1126/science.1074973. Consultado el 8 de marzo de 2023.
- ↑ Dekker, Job (Enero de 2006). «The three 'C' s of chromosome conformation capture: controls, controls, controls». Nature Methods 3 (1): 17-21. ISSN 1548-7091. PMID 16369547. doi:10.1038/nmeth823. Consultado el 8 de marzo de 2023.
- ↑ Tan, Longzhi; Xing, Dong; Chang, Chi-Han; Li, Heng; Xie, X. Sunney (31 de agosto de 2018). «Three-dimensional genome structures of single diploid human cells». Science (New York, N.Y.) 361 (6405): 924-928. ISSN 1095-9203. PMC 6360088. PMID 30166492. doi:10.1126/science.aat5641. Consultado el 8 de marzo de 2023.
Bibliografía
editar- Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Morgan, David; Raff, Martin; Roberts, Keith; Walter, Peter; Wilson, John ; Hunt, Tim (2010). «cap.4: DNA, cromosomos e genomas». Biologia Molecular da Célula (en portugués) (6ª edición). Artmed Editora.
- Luger, K.; Mäder, A.W.; Richmond, R.K.; Sargent, D.F.; Richmond, T.J. (18 de septiembre de 1997). «Crystal structure of the nucleosome core particle at 2.8 A resolution». Nature (en inglés) 389 (6648): 251-260. PMID 9305837. doi:10.1038/38444.
- Németh, A.; Längst, G. (1 de febrero de 2004). «Chromatin higher order structure: opening up chromatin for transcription». Briefings in functional genomics & proteomics (en inglés) 2 (4): 334-343. PMID 15163368. doi:10.1093/bfgp/2.4.334.
- Olins, D. E.; Olins, A. L. (1 de octubre de 2003). «Chromatin history: our view from the bridge». Nature reviews. Molecular cell biology (en inglés) 4 (10): 809-814. PMID 14570061. doi:10.1038/nrm1225.
