Vacunología inversa
Esto emplea técnica bioinformática, iniciada por Rino Rappuoli y utilizada por primera vez contra el meningococo del serogrupo B.[1][2] Desde entonces, se ha utilizado en varias otras vacunas bacterianas.[3]
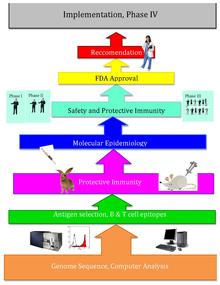
Enfoque computacional
editarLa idea básica detrás de la vacunación inversa es que se puede cribar un genoma patógeno completo utilizando enfoques bioinformáticos para encontrar genes. Algunos rasgos para los que se monitorean los genes pueden indicar antigenicidad e incluyen genes que codifican proteínas con localización extracelular, péptidos señal y epítopos de células B.[4] En el siguiente proceso esos genes se filtran en busca de atributos deseables que serían buenos objetivos para las vacunas, como las proteínas de la membrana externa. Una vez que se identifican los candidatos, se producen sintéticamente y se analizan en modelos animales de la infección.[5]
Historia
editarDespués de que Craig Venter publicara el genoma del primer organismo vivo en 1995, los genomas de otros microorganismos se volvieron más disponibles a lo largo del final del siglo XX. La vacunación inversa, el diseño de vacunas utilizando el genoma secuenciado del patógeno, surgió de esta nueva etapa y abundancia de información genómica, así como de los avances tecnológicos. La vacunación inversa es mucho más eficiente que la vacunación tradicional, que requiere el cultivo de grandes cantidades de microorganismos específicos, así como extensas pruebas de laboratorio húmedo.[cita requerida]
En el año 2000, Rino Rappuoli y el Instituto J. Craig Venter desarrollaron la primera vacuna utilizando la vacunación inversa contra el meningococo del serogrupo B. El Instituto J. Craig Venter y otros continuaron trabajando en vacunas para Estreptococo A, Estreptococo B, Estafilococo aureus y Estreptococo pneumoniae.[6]
Vacuna reversa con Meningococo B
editarLos intentos de vacunación inversa comenzaron con Meningococo B (MenB). El meningococo B causó más del 50% de la meningitis meningocócica y los científicos no fueron capaces de crear una vacuna exitosa para el patógeno debido a la estructura peculiar de la bacteria. La capa de polisacárido de esta bacteria es idéntica a la de un autoantígeno humano, pero sus proteínas de superficie varían mucho; y la falta de información sobre las proteínas de la superficie hizo que desarrollar una vacuna fuera extremadamente difícil. Como resultado, Rino Rappuoli y otros científicos recurrieron a la bioinformática para diseñar una vacuna funcional.[6]
Rappuoli y otros del Instituto J. Craig Venter secuenciaron por primera vez el genoma de MenB. Luego, escanearon el genoma secuenciado en busca de posibles antígenos. Encontraron más de 600 posibles antígenos, que fueron probados por factor de expresión en Escherichia coli. En los prototipos de vacunas se utilizaron los antígenos de aplicación más universales. Varias demostraron funcionar con éxito en ratones, sin embargo, estas proteínas por sí solas no interactuaron eficazmente con el sistema inmunológico humano debido a que no indujeron una buena respuesta inmunitaria para lograr la protección. Posteriormente, mediante la adición de vesículas de membrana externa que contienen lipopolisacáridos procedentes de la purificación de ampollas en cultivos gramnegativos. La adición de este auxiliar (previamente identificado mediante el uso de enfoques de vacunación convencionales) mejoró la respuesta inmune al nivel que se requería. Más tarde, se demostró que la vacuna era segura y eficaz en humanos adultos.[6]
Investigación posterior de vacunología inversa
editarDurante el desarrollo de la vacuna MenB, los científicos concluyeron que era seguro adoptar los mismos métodos de vacunación inversa para otros patógenos bacterianos. Las vacunas A Streptococcus y B Streptococcus fueron dos de las primeras vacunas inversas creadas. Debido a que esas cepas bacterianas inducen anticuerpos que reaccionan con antígenos humanos, las vacunas para esas bacterias debían no contener homologaciones con proteínas codificadas en el genoma humano para no causar reacciones adversas, estableciendo así la necesidad de una vacunación inversa basada en dicho genoma.[6]
Posteriormente, se utilizó la vacunación inversa para desarrollar vacunas para Estaphylococo aureus y Estreptococco pneumoniae resistentes a los antibióticos.[6]
Pros y contras
editarLa principal ventaja de la vacunación inversa es encontrar las reacciones objetivo de dichas vacunas de forma rápida y eficiente. Los métodos tradicionales pueden tardar décadas en desentrañar patógenos y antígenos, enfermedades e inmunidad. Sin embargo, In silico puede ser muy rápido, lo que permite identificar nuevas vacunas para probar en solo unos pocos años.[7] La desventaja es que solo las proteínas pueden dirigirse mediante este proceso. Considerando que, los enfoques de vacunología convencionales pueden encontrar otras dianas biomoleculares como los polisacáridos .
Software disponible
editarAunque el uso de tecnología bioinformática para desarrollar vacunas se ha convertido en algo habitual en los últimos diez años, una gran cantidad de laboratorios no cuentan con el software avanzado que puede hacer esto. Sin embargo, existe un número creciente de programas que hacen más accesible la información de vacunación inversa. NERVE es un programa de procesamiento de datos relativamente nuevo. Aunque debe descargarse y no incluye todas las predicciones de epítopos, ayuda a ahorrar algo de tiempo al combinar los pasos computacionales de la vacunación inversa en un solo programa. Vaxign, un programa aún más completo, se creó en 2008. Vaxign está basado en la web y es completamente de acceso público.[8]
Aunque se ha descubierto que Vaxign es extremadamente preciso y eficiente, algunos científicos todavía optan por utilizar el software en línea RANKPEP para las predicciones de enlaces de péptidos. Tanto Vaxign como RANKPEP emplean PSSM (Matrices de puntuación específicas de posición) al analizar secuencias de proteínas o alineaciones de secuencias.[9]
Los proyectos de bioinformática asistidos por computadora se están volviendo extremadamente populares, por su practicidad y nivel de exactitud, ya que ayudan a guiar los experimentos de laboratorio.[10]
Otros desarrollos debido a la vacunación inversa y la bioinformática
editar- La vacunación inversa ha provocado un mayor enfoque en la biología patógena.[6]
- La vacunación inversa condujo al descubrimiento de pilus en patógenos grampositivos como el estreptococo A, el estreptococo B y el neumococo. Anteriormente, se pensaba que todas las bacterias grampositivas no tenían pilus.
- La vacunación inversa también condujo al descubrimiento de la proteína de unión al factor G en el meningococo, que se une al factor H del complemento en los seres humanos. La unión al factor H del complemento permite que el meningococo crezca en la sangre humana mientras bloquea las vías alternativas. Este modelo no se ajusta a muchas especies animales, que no tienen el mismo factor de complemento H que los humanos, lo que indica la diferenciación del meningococo entre diferentes especies.
Referencias
editar- ↑ Pizza et al. Identification of Vaccine Candidates Against Serogroup B Meningococcus by Whole-Genome Sequencing Science 2000 287:1816-1820
- ↑ Martinón-Torres, Federico; Banzhoff, Angelika; Azzari, Chiara; Wals, Philippe De; Marlow, Robin; Marshall, Helen; Pizza, Mariagrazia; Rappuoli, Rino et al. (1 de julio de 2021). «Recent advances in meningococcal B disease prevention: real-world evidence from 4CMenB vaccination». Journal of Infection (en inglés) 83 (1): 17-26. ISSN 0163-4453. PMID 33933528. doi:10.1016/j.jinf.2021.04.031. Consultado el 16 de julio de 2021.
- ↑ Rappuoli, Rino. Reverse Vaccinology Current Opinion in Microbiology 2000, 3:445–450
- ↑ CH Woelk, et al. "Improving reverse vaccinology with a machine learning approach." Vaccine 29, no. 45 (n.d.): 8156-8164. Science Citation Index, EBSCOhost (accessed September 30, 2012).
- ↑ Michalik, M., Djahanshiri, B., Leo, J. C., & Linke, D. (2016). Reverse Vaccinology: The Pathway from Genomes and Epitope Predictions to Tailored Recombinant Vaccines. Methods in Molecular Biology, 1403, 87–106. https://doi.org/10.1007/978-1-4939-3387-7_4
- ↑ a b c d e f Alessandro S, Rino R. Review: Reverse Vaccinology: Developing Vaccines in the Era of Genomics. Immunity [serial online]. n.d.;33:530-541. Available from: ScienceDirect, Ipswich, MA. Accessed September 30, 2012.
- ↑ Rappuoli, R. & A. Aderem. 2011. A 2020 Vision for vaccines against HIV, tuberculosis and malaria. Nature 473: 463.
- ↑ He Y, Xiang Z, Mobley H. Vaxign: the first web-based vaccine design program for reverse vaccinology and applications for vaccine development. Journal of Biomedicine & Biotechnology [serial online]. 2010;Available from: CINAHL Plus with Full Text, Ipswich, MA. Accessed September 30, 2012.
- ↑ Reche PA, Glutting JP and Reinherz EL. Prediction of MHC Class I Binding Peptides Using Profile Motifs. Human Immunology 63, 701-709 (2002).
- ↑ Sandro V, Jennifer L. G, Francesco F, et al. Review: Computer-aided biotechnology: from immuno-informatics to reverse vaccinology. Trends In Biotechnology [serial online]. n.d.;26:190-200. Available from: ScienceDirect, Ipswich, MA. Accessed September 30, 2012.