Nucleocytoviricota
Nucleocytoviricota es un filo al que pertenece actualmente 13 familias de virus ADN según el ICTV y 1 familia está en proceso de incorporación. Son conocidos como virus nucleocitoplasmáticos de ADN de gran tamaño, virus gigantes, (acortado girus, de virus gigantes), VNCAGT o NCLDV por sus siglas en inglés. Los virus de este filo infectan principalmente protistas e invertebrados, unos muy pocos infectan vertebrados, entre ellos el ser humano, como el virus de la viruela.
| Nucleocytoviricota | ||
|---|---|---|
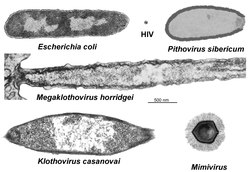 Comparación de los virus gigantes con la bacteria Escherichia coli y el virus del VIH. | ||
| Taxonomía | ||
| Dominio: | Varidnaviria | |
| Reino: | Bamfordvirae | |
| Filo: | Nucleocytoviricota | |
| Clasificación de Baltimore | ||
| Grupo: | I (Virus ADN bicatenario) | |
| Clasificación | ||
| Sinonimia | ||
| ||
Estos virus son tan grandes como las más pequeñas bacterias (o incluso mayores), tanto en la longitud de su ADN (que va desde 300 Kb a 2,5 Mb) como en diámetro (de 200 a 1000 nm). A modo de comparación, la bacteria de vida libre más pequeña, Mycoplasma genitalium, presenta 450 nm de diámetro y codifica únicamente 482 proteínas. En cambio el Mimivirus codifica 979 proteínas. Otro ejemplo de virus gigante es el Megavirus chilensis, que posee un genoma de alrededor de 1,26 Mb y un diámetro de alrededor de 700 nm. Los más grandes hallados hasta ahora son el Pandoravirus dulcis (con 1,9 mpb), encontrado en un lago en Australia y el Pandoravirus salinus (con 2,5 mpb), que llegan a medir aproximadamente 1 μm de diámetro.[2]
En 2019 se informó del descubrimiento de virus gigantes de un tamaño comparable al de bacterias comunes o incluso mayores y que se han identificado en los quetognatos. Estos virus pueden medir alrededor de 3,1 a 3,9 mpb. Se ha detectado la presencia de ribosomas, una característica ausente en los demás virus.[3][4][5] Las especies han sido nombradas como Megaklothovirus y Klothovirus, sin embargo, todavía falta más investigación sobre estos virus para conocer más detalles.
Características y razones para crear esta agrupación
editarTodas estas familias tienen en común el tamaño de su ADN, el poseer genes poco habituales en el resto de virus y la estructura del virión y del ADN, todos bicatenarios.
Una característica que los distingue radicalmente del resto de virus es su abertura en estrella, visible después de liberar su carga genética en el huésped. La salida del genoma del virus se realiza por esta hendidura, mientras que la entrada del ADN, en el ensamblaje intracelular del virus, se realiza exclusivamente por el lado opuesto. Son los únicos virus que poseen un lado de salida y un lado de entrada del ADN determinados.[6]
Se conocen 47 genes del core de los virus gigantes. Se incluyen los 4 genes clave involucrados en la replicación y reparación del ADN: ADN polimerasa B, topoisomerasa II A, Flap endonucleasa y el factor antígeno nuclear de proliferación celular. Otros genes codifican proteínas, la ARN polimerasa dependiente de ADN II y el factor de transcripción II B.
Igualmente se ha descubierto que, además, muchos de ellos son la diana de diferentes tipos de virófagos (virus que parasitan otros virus).
Todos los análisis filogenéticos y estructurales sugieren que los virus gigantes son un grupo monofilético, por lo que descienden de un solo virus ancestral.
Los virus gigantes parecen haber evolucionado de virus pequeños clásicos del filo Preplasmiviricota, que aumentaron su genoma y el tamaño del virión mediante la duplicación y deleción de genes, la inclusión de elementos genéticos móviles y la adquisición masiva de genes de huéspedes y bacterias, incluidos los genes para la traducción y los genes informáticos que se consideran los más resistentes a la transferencia horizontal.[7][8][9]
Relación con los organismos celulares y otros virus
editarEl genoma ancestral era complejo, con al menos 47 genes, incluyendo: (1) la maquinaria de replicación, (2) hasta 4 subunidades de ARN polimerasa, (3) al menos tres factores de transcripción, (4) enzimas de poliadenilación y colocación de la caperuza, (5) el sistema de empaquetamiento de ADN, (6) genes codificadores de proteínas, (7) componentes estructurales de la cápside y la envoltura vírica.[10] Se ha detectado que estos virus también poseen en sus genomas elementos genéticos móviles como transposones únicos llamados transpoviriones y plásmidos citoplasmáticos.[11] Estos elementos se consideraban propios de los organismos celulares. Además de genes relacionados con el funcionamiento del metabolismo, la traducción y genes informáticos que hasta hace poco se consideraban propios de los organismos celulares. En los recientes virus gigantes de quetognatos se ha informado de la presencia de ribosomas.[3] Por ello se ha afirmado que los virus gigantes pueden pertenecer universalmente al árbol filogenético de la vida.[12][10]
Se ha sugerido que el origen de los virus gigantes es anterior al de los eucariotas o incluso anterior al del último antepasado común universal (LUCA) dado que se han detectado algunos genes compartidos únicamente entre los virus gigantes, las bacterias y las arqueas, lo que implica que el ancestro de los virus gigantes habría infectado a los procariotas en el pasado.[13][10] Las proteínas de la cápside y las ARN polimerasas no tienen homología con los organismos celulares.[14] Análisis filogenéticos sugieren que dos ARN polimerasas dependientes de ADN eucariotas se originaron de las que portan los virus gigantes.[15] Además, se ha propuesto que los virus gigantes pudieron haber originado el núcleo de las células eucariotas al haberse incorporado el virus dentro de la célula donde en lugar de replicarse y destruir la célula huésped, permanecería dentro de la célula originando posteriormente el núcleo y dando lugar a otras innovaciones genómicas. Esta teoría es conocida como la "eucariogénesis viral".[16] Las proteínas de la cápside de los virus gigantes son en rollo de gelatina vertical por lo que se les incluye en el dominio Varidnaviria que también contiene a los adenovirus, los virófagos y varias familias de virus procariotas, ya que se cree que ambos comparten un ancestro en común que se habría originado antes que el último antepasado común universal (LUCA).[17] Dentro de este dominio se ha sugerido que los virus gigantes pudieron dar origen a los virus de la clase Naldaviricetes por reducción o descender de un ancestro compartido.[18] Algunos genes y proteínas exclusivas de los virus gigantes también se han conservado en una gran variedad de virus que infectan organismos de todos los dominios (Eukarya, Archaea y Bacteria). Esto hace suponer que el ancestro de los virus gigantes está relacionado con un tipo de virus de ADN que existió antes que los organismos celulares y jugó un papel esencial en el desarrollo de la vida en la Tierra.[19][20]
Debido a que comparten varias características con los organismos celulares, se han hecho intentos por incluir a este grupo de virus en el árbol filogenético de la vida.[10] Sin embargo, este argumento se ve desafiado por la transferencia horizontal de genes, ya que se han revelado constantes transferencias horizontales entre los virus gigantes y sus huéspedes eucariotas.[12] Algunos autores sostienen que la inclusión de los virus gigantes u otros virus en el árbol filogenético de la vida es irrelevante.[21]
Recientemente, se han propuesto dos filogenias para los virus gigantes con respecto a los organismos celulares.
Basada en la secuencia de aminoácidos y genes, esta filogenia propone que los virus gigantes serían probablemente un grupo monofilético, siendo las relaciones filogenéticas entre subgrupos las siguientes.[22][23]
| Biota |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Otros estudios sobre los proteomas y los pliegues de las proteínas víricas concluyen que los virus gigantes, junto con los restantes virus, formarían un grupo complejamente parafilético. El análisis sque los virus se habrían originado durante el mundo de ARN en protobiontes que posteriormente se separaran para dar origen a los procariotas.[24] Según esta hipótesis, los mimivirus constituirían el grupo hermano de los organismos celulares, mientras que las demás familias de virus gigantes se habrían ramificado de forma temprana y parafilética antes de los mimivirus y los organismos celulares.[25] Lo mismo sucedería con las demás familias de virus ADN y ARN que se bifurcaban en manera parafilética y mezclada antes de los virus gigantes y los organismos celulares.[26][27][28]
| Biota |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Una posible agrupación de Cytota parientes acelulares cercanos se incluiría en Cytotamorpha.
Véase también
editarReferencias
editar- ↑ Claire Bertelli, Linda Mueller, Vincent Thomas, Trestan Pillonel, Nicolas Jacquier, Gilbert Greub (2017). Cedratvirus lausannensis – digging into Pithoviridae diversity. Online Library.
- ↑ N. Philippe; M. Legendre; G. Doutre; Y. Couté; O. Poirot; M. Lescot; D. Arslan; V. Seltzer; L. Bertaux; C. Bruley; J. Garin; J.-M. Claverie; C. Abergel N. Pandoraviruses: Amoeba Viruses with Genomes up to 2,5 Mb Reaching That of Parasitic Eukaryotes. Science (2013). DOI: 10.1126/science.1239181.
- ↑ a b Giant gene thieves
- ↑ https://www.hilarispublisher.com/open-access/serendipitous-discovery-in-a-marine-invertebrate-phylum-chaetognatha-of-the-longest-giant-viruses-reported-to-date.pdf
- ↑ Shinn GL, Bullard BL (19 de septiembre de 2018). «Ultrastructure of Meelsvirus: A nuclear virus of arrow worms (phylum Chaetognatha) producing giant "tailed" virions». En San Martin, Carmen, ed. PLOS ONE 13 (9): e0203282. Bibcode:2018PLoSO..1303282S. PMC 6145532. PMID 30231047. doi:10.1371/journal.pone.0203282.
- ↑ Virus gigantes L. van Etten, James (2012). Investigación y Ciencia, ed. Virus gigantes.
- ↑ David Moreira, Céline Brochier-Armanet (2008). Giant viruses, giant chimeras: The multiple evolutionary histories of Mimivirus genes. NCBI.
- ↑ Eugene V. Koonin, Natalya Yutin (2019). Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism. Science Direct.
- ↑ Jonathan Filée (2013). Route of NCLDV evolution: the genomic accordion. Science Direct.
- ↑ a b c d Mickaël Boyer, Mohammed-Amine Madoui, Gregory Gimenez, Bernard La Scola, Didier Raoult (2010). Phylogenetic and Phyletic Studies of Informational Genes in Genomes Highlight Existence of a 4th Domain of Life Including Giant Viruses. Plos One. https://doi.org/10.1371/journal.pone.0015530
- ↑ Eugene Koonin, Valerian V Doljja (2014). Virus World as an Evolutionary Network of Viruses and Capsidless Selfish Elements. Microbiology and Molecular Biology Reviews.
- ↑ a b Tom Williams, T Martin Embley, Eva Heinz (2011). Informational Gene Phylogenies Do Not Support a Fourth Domain of Life for Nucleocytoplasmic Large DNA Viruses. Researchgate.
- ↑ Didier Raoult et al (2004). The 1.2-Megabase Genome Sequence of Mimivirus. Science Direct.
- ↑ Arshan Nasir, Kyung Mo Kim, Gustavo Caetano-Anolles (2012). Giant viruses coexisted with the cellular ancestors and represent a distinct supergroup along with superkingdoms Archaea, Bacteria and Eukarya. BMC Evolutionary Biology.
- ↑ Guglielmini, Julien; Woo, Anthony C.; Krupovic, Mart; Forterre, Patrick; Gaia, Morgan (10 de septiembre de 2019). «Diversification of giant and large eukaryotic dsDNA viruses predated the origin of modern eukaryotes». Proceedings of the National Academy of Sciences (en inglés) 116 (39): 19585-19592. ISSN 0027-8424. PMC 6765235. PMID 31506349. doi:10.1073/pnas.1912006116.
- ↑ Philip John Livingstone Bell (2001). «Viral eukaryogenesis: Was the ancestor of the nucleus a complex DNA virus?». Journal of Molecular Evolution 53 (3): 251-256. Bibcode:2001JMolE..53..251L. PMID 11523012. doi:10.1007/s002390010215.
- ↑ Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (18 de octubre de 2019). «Create a megataxonomic framework, filling all principal taxonomic ranks, for DNA viruses encoding vertical jelly roll-type major capsid proteins» (docx). International Committee on Taxonomy of Viruses (en inglés). Consultado el 10 de junio de 2020.
- ↑ Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic (2015). Origins and evolution of viruses of eukaryotes: The ultimate modularity. Sciences Direct.
- ↑ Eugene Koonin, Valerian V Dolja (2014). A virocentric perspective on the evolution of life. Science Direct.
- ↑ Forterre, Patrick (2006). «Three RNA cells for ribosomal lineages and three DNA viruses to replicate their genomes: A hypothesis for the origin of cellular domain». PNAS 106 (10): 3669-3674. PMC 1450140. PMID 16505372. doi:10.1073/pnas.0510333103.
- ↑ David Moreira, Purificación-Lopéz García (2009). Ten reasons to exclude viruses from the tree of life. Nature.
- ↑ Vikas Sharma, Philippe Colson, Olivier Chabrol, Pierre Pontarotti, Didier Raoult (2015). Pithovirus sibericum, a new bona fide member of the “Fourth TRUC” club. Frontiers. https://doi.org/10.3389/fmicb.2015.00722
- ↑ Philippe Colson, Anthony Levasseur, Bernard La Scola, Vikas Sharma, Gustavo Caetano Anollés, Pierre Pontarotti, Didier Raoult (2018). Ancestrality and Mosaicism of Giant Viruses Supporting the Definition of the Fourth TRUC of Microbes. Frontiers. https://doi.org/10.3389/fmicb.2018.02668
- ↑ Viral evolution: Primordial cellular origins and late adaptation to parasitism. NCBI.
- ↑ Rijja Hussain Bokhari, Nooreen Amirjan, Hyeonsoo Jeong, Kyung Mo Kim, Gustavo Caetano-Anollés, Arshan Nasir (2020). Bacterial Origin and Reductive Evolution of the CPR Group. Oxford Academic. https://doi.org/10.1093/gbe/evaa024
- ↑ The distribution and impact of viral lineages in domains of life. Frontiers.
- ↑ Do Viruses Exchange Genes across Superkingdoms of Life?. Frontiers.
- ↑ Arshan, Nasir; Caetano-Anollés, Gustavo (25 de septiembre de 2015). «A phylogenomic data-driven exploration of viral origins and evolution». Science Advances 1 (8): e1500527. Bibcode:2015SciA....1E0527N. PMC 4643759. PMID 26601271. doi:10.1126/sciadv.1500527.