CRISPR/Cas
La edición génica o genética CRISPR (pronunciado /ˈkrɪspər/ «crisper»), acrónimo de «Clustered Regularly Interspaced Short Palindromic Repeats» (en español: repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas), es una técnica de ingeniería genética en biología molecular mediante la cual se pueden modificar los genomas de organismos vivos. Se basa en una versión simplificada del sistema de defensa antiviral bacteriano CRISPR-Cas9. Al introducir en una célula la nucleasa Cas9 en complejo con un ARN guía sintético (ARNg), se puede cortar el genoma de la célula en el lugar deseado, lo que permite eliminar genes existentes y/o añadir otros nuevos in vivo.[1]
La técnica se considera muy importante en biotecnología y medicina, ya que permite editar genomas in vivo de forma muy precisa y sencilla. Puede utilizarse en la creación de nuevos medicamentos, productos agrícolas y organismos genéticamente modificados, o como medio de control de patógenos y plagas. También tiene posibilidades en el tratamiento de enfermedades genéticas hereditarias, así como de enfermedades derivadas de mutaciones somáticas, como el cáncer. Sin embargo, su uso en la modificación genética de la línea germinal humana es muy controvertido. El desarrollo de la técnica les valió a Jennifer Doudna y Emmanuelle Charpentier el Premio Nobel de Química en 2020.[2][3] El tercer grupo de investigadores que compartió el Premio Kavli por el mismo descubrimiento,[4] liderado por Virginijus Šikšnys, no fue galardonado con el Nobel.[5][6][7]
Funcionando como unas tijeras genéticas, la nucleasa Cas9 abre las dos hebras de la secuencia de ADN objetivo para introducir la modificación por uno de dos métodos. Las mutaciones knock-in, facilitadas por la reparación dirigida por homología (HDR), son la vía tradicional de los métodos de edición genómica dirigida.[1] Esto permite la introducción de daños en el ADN y su reparación. La HDR emplea el uso de secuencias de ADN similares para impulsar la reparación de la rotura mediante la incorporación de ADN exógeno para que funcione como plantilla de reparación.[1] Este método se basa en la aparición periódica y aislada de daños en el ADN en el lugar objetivo para que comience la reparación. Las mutaciones knock-out causadas por CRISPR-Cas9 son el resultado de la reparación de la rotura de doble cadena mediante la unión de extremos no homólogos (NHEJ) o la unión de extremos mediada por POLQ/polimerasa theta (TMEJ). Estas vías de unión final pueden dar lugar a menudo a deleciones o inserciones aleatorias en el lugar de reparación, que pueden interrumpir o alterar la funcionalidad de los genes. Por lo tanto, la ingeniería genómica mediante CRISPR-Cas9 ofrece a los investigadores la posibilidad de alterar genes de forma aleatoria y selectiva.
Aunque la edición del genoma en células eucariotas ha sido posible mediante diversos métodos desde la década de 1980, los métodos empleados habían demostrado ser ineficaces y poco prácticos de aplicar a gran escala. Con el descubrimiento de las CRISPR y, en concreto, de la molécula nucleasa Cas9, se hizo posible una edición eficiente y altamente selectiva. La Cas9, derivada de la especie bacteriana Streptococcus pyogenes, ha facilitado la modificación genómica selectiva en células eucariotas al permitir un método fiable para crear una rotura selectiva en un lugar específico designado por las cadenas guía ARNcr y ARNtracr.[8] La facilidad con la que los investigadores pueden insertar Cas9 y ARN molde para silenciar o causar mutaciones puntuales en loci específicos ha demostrado ser inestimable para el mapeo rápido y eficiente de modelos genómicos y procesos biológicos asociados a diversos genes en una variedad de eucariotas. Se han desarrollado nuevas variantes de la nucleasa Cas9 que reducen significativamente la actividad fuera del objetivo.[9]
Las técnicas de edición del genoma CRISPR-Cas9 tienen muchas aplicaciones potenciales. El uso del complejo CRISPR-Cas9-ARNg para la edición del genoma[10] fue elegido por la AAAS como avance del año en 2015.[11] Se han planteado muchas preocupaciones bioéticas ante la perspectiva de utilizar las CRISPR para la edición de la línea germinal, especialmente en embriones humanos.[12] En 2023, se aprobó en el Reino Unido el uso de Casgevy, el primer fármaco que utiliza la edición genética CRISPR para curar la anemia falciforme y la beta talasemia.[13][14] La Administración de Alimentos y Medicamentos aprobó el uso de Casgevy en Estados Unidos el 8 de diciembre de 2023.[15]
Historia
editarOtros métodos
editarA principios de la década de 2000, investigadores alemanes comenzaron a desarrollar nucleasas con dedos de zinc (ZFN), proteínas sintéticas cuyos dominios de unión al ADN les permiten crear rupturas de doble hebra en el ADN en puntos específicos. Las ZFN tienen una mayor precisión y la ventaja de ser más pequeñas que Cas9, pero las ZFN no se usan tan comúnmente como los métodos basados en las CRISPR. En 2010, las nucleasas sintéticas llamadas nucleasas efectoras tipo activador de transcripción (TALEN) proporcionaron una forma más sencilla de dirigir una ruptura de doble hebra a una ubicación específica en la cadena de ADN. Tanto las nucleasas con dedo de zinc como las TALEN requieren el diseño y creación de una proteína personalizada para cada secuencia de ADN objetivo, lo cual es un proceso mucho más difícil y que consume más tiempo que diseñar ARN guías. Las CRISPR son mucho más fáciles de diseñar porque el proceso solo requiere sintetizar una secuencia corta de ARN, un procedimiento que ya se utiliza ampliamente para muchas otras técnicas de biología molecular (por ejemplo, crear cebadores de oligonucleótidos).[16]
Mientras que métodos como la interferencia por ARN (ARNi) no suprimen completamente la función génica, CRISPR, ZFN y TALEN proporcionan una inactivación génica completa e irreversible.[17] Las CRISPR también puede dirigirse a varios sitios de ADN simultáneamente simplemente introduciendo diferentes ARN guías (ARNg). Además, los costos de emplear CRISPR son relativamente bajos.[17][18][19]
Descubrimiento
editarEn 2005, Alexander Bolotin, del Instituto Nacional de Investigación Agronómica de Francia (INRA), descubrió unas locus CRISPR que contenían genes Cas nuevos, destacando uno que codificaba una gran proteína conocida como Cas9.[20]
En 2006, Eugene Koonin, del Centro Nacional para la Información Biotecnológica de Estados Unidos, NIH, propuso una explicación sobre cómo las cascadas CRISPR funcionan como un sistema inmunitario bacteriano.[20]
En 2007, Philippe Horvath, de Danisco France SAS, demostró experimentalmente cómo los sistemas CRISPR son un sistema inmunitario adaptativo e integran nuevo ADN de fagos en la matriz CRISPR, lo cual es cómo combaten la siguiente ola de fagos atacantes.[20]
En 2012, el equipo de investigación dirigido por Jennifer Doudna, profesora de la Universidad de California en Berkeley, y Emmanuelle Charpentier, profesora de la Universidad de Umeå, fueron las primeras personas en identificar, divulgar y presentar una solicitud de patente para el sistema CRISPR-Cas9 necesario para editar el ADN.[20] También publicaron su descubrimiento de que CRISPR-Cas9 podría programarse con ARN para editar el ADN genómico, ahora considerado uno de los descubrimientos más significativos en la historia de la biología.
Patentes y comercialización
editarEn noviembre de 2013, SAGE Labs (parte del grupo Horizon Discovery) tenía derechos exclusivos de una de esas compañías para producir y vender ratas genéticamente modificadas y derechos no exclusivos para modelos de ratones y conejos.[21] Para 2015, Thermo Fisher Scientific había licenciado propiedad intelectual de ToolGen para desarrollar kits de reactivos CRISPR.[22]
En diciembre de 2014, los derechos de patente de las CRISPR estaban en disputa. Varias compañías se formaron para desarrollar medicamentos y herramientas de investigación relacionadas.[23] A medida que las empresas aumentaban la financiación, surgieron dudas sobre si las CRISPR podrían monetizarse rápidamente.[24] En 2014, Feng Zhang del Instituto Broad del MIT y Harvard y otros nueve fueron galardonados con la patente estadounidense número 8,697,359[25] sobre el uso de la edición genética CRISPR-Cas9 en eucariotas. Aunque Charpentier y Doudna (referidas como CVC) fueron acreditadas por la concepción de las CRISPR, el Instituto Broad fue el primero en lograr una "reducción a la práctica" según los jueces de patentes Sally Gardner Lane, James T. Moore y Deborah Katz.[26]
El primer conjunto de patentes se otorgó al equipo de Broad en 2015, lo que llevó a los abogados del grupo CVC a solicitar el primer procedimiento de interferencia.[27] En febrero de 2017, la Oficina de Patentes de EE. UU. falló en un caso de interferencia de patentes presentado por la Universidad de California con respecto a las patentes emitidas al Instituto Broad, y encontró que las patentes de Broad, con reclamos que cubren la aplicación de CRISPR-Cas9 en células eucariotas, eran distintas de las invenciones reclamadas por la Universidad de California.[28][29][30]
Poco después, la Universidad de California presentó una apelación a esta decisión.[31][32] En 2019 se abrió la segunda disputa de interferencia. Esto fue en respuesta a las solicitudes de patente realizadas por CVC que requerían que la junta de apelaciones determinara el inventor original de la tecnología. La USPTO falló en marzo de 2022 en contra de UC, afirmando que el Instituto Broad fue el primero en presentar. La decisión afectó muchos de los acuerdos de licencia para la tecnología de edición CRISPR que fue licenciada de UC Berkeley. UC declaró su intención de apelar el fallo de la USPTO.[33]
Acontecimientos recientes
editarEn marzo de 2017, la Oficina Europea de Patentes (OEP) anunció su intención de permitir reivindicaciones para editar todo tipo de células al Instituto Max-Planck de Berlín, la Universidad de California y la Universidad de Viena,[34][35] y en agosto de 2017, la OEP anunció su intención de permitir reivindicaciones CRISPR en una solicitud de patente que MilliporeSigma había presentado.[34] En agosto de 2017, la situación de las patentes en Europa era compleja, con MilliporeSigma, ToolGen, la Universidad de Vilnius y Harvard contendiendo por reivindicaciones, junto con la Universidad de California y Broad.[36]
En julio de 2018, el Tribunal de Justicia de la Unión Europea dictaminó que la edición genética para plantas era una subcategoría de los alimentos OGM y, por tanto, que la técnica de las CRISPR se regularía en adelante en la Unión Europea por sus normas y reglamentos para los OGM.[37]
En febrero de 2020, una investigación en Estados Unidos demostró la seguridad de la edición genética CRISPR en tres pacientes con cáncer.[38]
En octubre de 2020, las investigadoras Emmanuelle Charpentier y Jennifer Doudna recibieron el Premio Nobel de Química por su trabajo en este campo.[39][40] Ellas hicieron historia al ser las dos primeras mujeres en compartir este galardón sin un colaborador masculino.[5][41]
En junio de 2021, el primer pequeño ensayo clínico de edición génica CRISPR intravenosa en humanos se concluyó con resultados prometedores [42][43]
En septiembre de 2021, el primer alimento editado con CRISPR salió a la venta en Japón. Los tomates se modificaron genéticamente para obtener una cantidad de GABA cinco veces superior a la normal.[44][45] Las CRISPR se aplicaron por primera vez en tomates en 2014.[46]
En diciembre de 2021, se informó de que el primer animal marino/marisco modificado genéticamente con las CRISPR y el segundo grupo de alimentos modificados con las CRISPR habían salido a la venta en Japón: dos peces de los cuales una especie crece el doble de tamaño que los especímenes naturales debido a la alteración de la leptina, que controla el apetito, y el otro crece hasta 1,2 veces el tamaño medio natural con la misma cantidad de comida debido a la miostatina desactivada, que inhibe el crecimiento muscular.[47][48][49]
Un estudio de 2022 ha descubierto que saber más sobre los tomates CRISPR tuvo un fuerte efecto en la preferencia de los participantes. «Casi la mitad de los 32 participantes de Alemania que son científicos demostraron elecciones constantes, mientras que la mayoría mostró una mayor disposición a comprar tomates CRISPR, en su mayoría no científicos».[50][51][52]
La UC Berkeley anunció en mayo de 2021 su intención de subastar tokens no fungibles tanto de la patente de edición genética CRISPR como de la inmunoterapia contra el cáncer. No obstante, en este caso la universidad conservaría la propiedad de las patentes.[53][54] El 85 % de los fondos recaudados con la venta de la colección denominada The Fourth Pillar se destinaría a financiar la investigación.[55][56] Se vendió en junio de 2022 por 22 éteres, que en aquel momento eran unos 54.000 dólares.[57]
En noviembre de 2023, la Agencia Reguladora de Medicamentos y Productos Sanitarios del Reino Unido (MHRA) se convirtió en la primera del mundo en aprobar el uso del primer medicamento basado en la edición genética CRISPR, Casgevy, para tratar la anemia falciforme y la beta talasemia. Casgevy, o autotemcel exagamglogénico, actúa directamente sobre los genes de las células madre del interior de los huesos del paciente, haciéndolas producir glóbulos rojos sanos. Este tratamiento evita así la necesidad de transfusiones de sangre periódicas y costosas.[13][14]
En diciembre de 2023, la FDA aprobó la primera terapia génica en EE.UU. para tratar a pacientes con anemia falciforme (ECF). La FDA aprobó dos tratamientos emblemáticos, Casgevy y Lyfgenia, que representan las primeras terapias génicas celulares para el tratamiento de la ECF.[58]
Ingeniería genómica
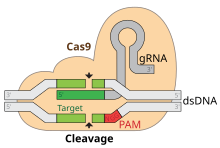
editarLa edición genómica CRISPR-Cas9 se lleva a cabo con un sistema CRISPR de tipo II. Cuando se utiliza para la edición del genoma, este sistema incluye una ribonucleoproteína (RNP), formada por Cas9, ARNcr y ARNtracr, junto con una plantilla opcional de reparación del ADN.
Componentes principales
editar| Componente | Función |
|---|---|
| ARNcr | Contiene el ARN guía que localiza el segmento correcto del ADN del huésped junto con una región que se une a ARNtracr (generalmente en forma de tallo-bucle), formando un complejo activo. |
| ARN | Se une a ARNcr y forma un complejo activo. |
| ARNgu | Los ARN guía únicos son un ARN combinado que consiste en un ARNtracr y al menos un ARNcr. |
| Cas9 (más comúnmente) | Una enzima cuya forma activa es capaz de modificar el ADN. Existen muchas variantes con diferentes funciones (es decir, corte de una sola hebra, corte de doble hebra, unida al ADN) debido a la función de reconocimiento del sitio de ADN de cada enzima. |
| Plantilla de reparación | Molécula de ADN utilizada como plantilla en el proceso de reparación del ADN de la célula huésped, permitiendo la inserción de una secuencia específica de ADN en el segmento roto del huésped por Cas9. |
Las CRISPR-Cas9 a menudo emplean plásmidos, que codifican los componentes RNP, para transfectar las células objetivo, o el RNP se ensambla antes de añadirse a las células mediante nucleofección.[59] Los principales componentes de este plásmido se muestran en la imagen y se enumeran en la tabla. El ARNcr está diseñado específicamente para cada aplicación, ya que esta es la secuencia que Cas9 utiliza para identificar y unirse directamente a secuencias específicas dentro del ADN de la célula huésped. El ARNcr debe unirse solo donde se desea la edición. La plantilla de reparación también se diseña específicamente para cada aplicación, ya que debe complementar en cierta medida las secuencias de ADN a ambos lados del corte y también contener cualquier secuencia que se desee insertar en el genoma del huésped.
Múltiples ARNcr y el ARNtracr pueden empaquetarse juntos para formar un ARN guía único (ARNgu).[60] Este ARNgu puede incluirse junto con el gen que codifica la proteína Cas9 y hacerse en un plásmido para ser transfectado en las células. Hay muchas herramientas en línea disponibles para ayudar en el diseño de secuencias efectivas de ARNgu.[61][62]
Alternativas a Cas9
editarLas proteínas alternativas a Cas9 incluyen las siguientes:
| Proteína | Características y uso principal | Año |
|---|---|---|
| Cas12 | Cas12a es más pequeña y simple que Cas9; Cas12b, entre otros usos, para la ingeniería del genoma de plantas.[63][64] | |
| Cas13 | Para la edición de ARN.[65] | |
| Cas3[66][67] | Crea una brecha amplia de una sola hebra.[68] | 2019 |
| CasMINI | Aproximadamente el doble de compacta que las más comúnmente utilizadas Cas9 y Cas12a.[69][70] | 2021 |
| SuperFi-Cas9 | Más precisa sin una disminución en la velocidad.[71][72] | 2022 |
| Cas7-11 | Edición de ARN.[73] | 2022 |
| Reparación de ADN con una plantilla de cromosomas | Este método solo es aplicable a organismos cuyo cromosoma coincidente tiene los genes deseados.
|
2022 |
Estructura
editarLas CRISPR-Cas9 ofrecen un alto grado de fidelidad y una construcción relativamente simple. Depende de dos factores para su especificidad: la secuencia objetivo y la secuencia del motivo adyacente de protoespaciador (PAM). La secuencia objetivo tiene 20 bases de longitud como parte de cada locus de CRISPR en la matriz de ARNcr.[59] Una matriz típica de ARNcr tiene múltiples secuencias objetivo únicas. Las proteínas Cas9 seleccionan la ubicación correcta en el genoma del huésped utilizando la secuencia para unirse con pares de bases en el ADN del huésped. La secuencia no forma parte de la proteína Cas9 y, como resultado, es personalizable y puede sintetizarse de forma independiente.[76][77]
La secuencia PAM en el genoma del huésped es reconocida por Cas9. El Cas9 no puede modificarse fácilmente para reconocer una secuencia PAM diferente. Sin embargo, esto no es demasiado limitante, ya que generalmente es una secuencia muy corta y no específica que ocurre frecuentemente en muchos lugares a lo largo del genoma (por ejemplo, la secuencia PAM de SpCas9 es 5'-NGG-3' y en el genoma humano ocurre aproximadamente cada 8 a 12 pares de bases).[59]
Una vez que estas secuencias se han ensamblado en un plásmido y transfectado en las células, la proteína Cas9, con la ayuda del ARNcr, encuentra la secuencia correcta en el ADN de la célula huésped y, dependiendo de la variante de Cas9, crea una ruptura de hebra sencilla o doble en la ubicación apropiada del ADN.[78]
Las rupturas de hebra sencilla adecuadamente espaciadas en el ADN del huésped pueden desencadenar la reparación dirigida por homología, que es menos propensa a errores que la unión de extremos no homóloga que típicamente sigue a una ruptura de doble hebra. Proporcionar una plantilla de reparación de ADN permite la inserción de una secuencia específica de ADN en una ubicación exacta dentro del genoma. La plantilla de reparación debe extenderse de 40 a 90 pares de bases más allá de la ruptura de ADN inducida por Cas9.[59] El objetivo es que el proceso HDR nativo de la célula utilice la plantilla de reparación proporcionada e incorpore así la nueva secuencia en el genoma. Una vez incorporada, esta nueva secuencia es ahora parte del material genético de la célula y se transmite a sus células hijas. La inhibición transitoria combinada de NHEJ y TMEJ mediante una pequeña molécula y siRNAs puede aumentar la eficiencia de HDR hasta un 93% y simultáneamente prevenir la edición fuera del objetivo.[79]
Entrega
editarLa entrega de Cas9, ARNgu y complejos asociados en las células puede ocurrir mediante sistemas virales y no virales. La electroporación de ADN, ARN o ribonucleocomplejos es una técnica común, aunque puede resultar en efectos dañinos para las células objetivo.[80] También se han utilizado técnicas de transfección química que emplean lípidos y péptidos para introducir ARNgus en complejo con Cas9 en las células.[81][82] La entrega basada en nanopartículas también se ha utilizado para la transfección.[83] Los tipos de células que son más difíciles de transfectar (por ejemplo, células madre, neuronas y células hematopoyéticas) requieren sistemas de entrega más eficientes, como aquellos basados en lentivirus (LVs), adenovirus (AdV) y virus adenoasociados (AAV).[84][85][86]
Se ha encontrado que la eficiencia de las CRISPR-Cas9 aumenta significativamente cuando varios componentes del sistema, incluida toda la estructura de las CRISPR/Cas9, se entregan en forma ensamblada en lugar de usar transgénicos.[87][88] Esto ha encontrado un valor particular en cosechas genéticamente modificados para la comercialización masiva.[89][90] Dado que la maquinaria de replicación del huésped no es necesaria para producir estas proteínas, la posibilidad de reconocer la secuencia del ARNgu es casi nula, disminuyendo la probabilidad de efectos fuera del objetivo.[83]
Edición controlada del genoma
editarLas mejoras y variantes del sistema CRISPR-Cas9 se han centrado en introducir más control en su uso. Específicamente, la investigación dirigida a mejorar este sistema incluye mejorar su especificidad, su eficiencia y la granularidad de su poder de edición. Las técnicas pueden dividirse y clasificarse por el componente del sistema que modifican. Estos incluyen el uso de variantes diferentes o creaciones novedosas de la proteína Cas, el uso de una proteína efectora completamente diferente, la modificación del ARNgu o el uso de un enfoque algorítmico para identificar soluciones óptimas existentes.
La especificidad es un aspecto importante para mejorar el sistema CRISPR-Cas9 porque los efectos fuera del objetivo que genera tienen consecuencias graves para el genoma de la célula e invocan precaución en su uso. Minimizar los efectos fuera del objetivo es, por lo tanto, maximizar la seguridad del sistema. Las variaciones novedosas de las proteínas Cas9 que aumentan la especificidad incluyen proteínas efectoras con eficiencia y especificidad comparables a la SpCas9 original que son capaces de apuntar a las secuencias previamente inalcanzables y una variante que prácticamente no tiene mutaciones fuera del objetivo.[91][92] También se ha investigado en la ingeniería de nuevas proteínas Cas9, incluidas algunas que reemplazan parcialmente los nucleótidos de ARN en el ARNcr con ADN y un procedimiento de generación de mutantes de Cas9 guiado por la estructura que tuvo efectos fuera del objetivo reducidos.[93][94] Se ha demostrado que los ARNgu truncados iterativamente y los ARNg altamente estabilizados también disminuyen los efectos fuera del objetivo.[95][96] Los métodos computacionales, incluido el aprendizaje automático, se han utilizado para predecir la afinidad y crear secuencias únicas para el sistema para maximizar la especificidad para objetivos determinados.[97][98]
Varias variantes de CRISPR-Cas9 permiten la activación de genes o la edición del genoma con un desencadenante externo, como luz o pequeñas moléculas.[99][100][101] Estos incluyen sistemas CRISPR fotoactivables desarrollados fusionando socios proteicos sensibles a la luz con un dominio activador y un dCas9 para la activación de genes,[102][103] o fusionando dominios similares sensibles a la luz con dos constructos de Cas9 dividida,[104][105] o incorporando aminoácidos no naturales encapsulados en Cas9,[106] o modificando los ARN guía con complementos fotoescindibles para la edición del genoma.[107]
Los métodos para controlar la edición del genoma con pequeñas moléculas incluyen un Cas9 alostérico, sin edición de fondo detectable, que activará la unión y la escisión tras la adición de 4-hidroxitamoxifeno (4-HT),[99] inteína fusionada con Cas9 sensible a 4-HT,[108] o un Cas9 que es sensible a 4-HT cuando se fusiona con cuatro dominios ERT2.[109] La Cas9 dividida inducible por inteína permite la dimerización de fragmentos de Cas9[110] y el sistema de Cas9 dividida inducible por rapamicina desarrollado fusionando dos constructos de Cas9 dividida con fragmentos FRB y FKBP.[111] Otros estudios han podido inducir la transcripción de Cas9 con una pequeña molécula, doxiciclina.[112][113] Las pequeñas moléculas también se pueden usar para mejorar la reparación dirigida por homología,[114] a menudo inhibiendo la vía de unión de extremos no homólogos y/o la vía de unión de extremos mediados por theta.[115][116] Se creó un sistema con la proteína efectora Cpf1 que es inducido por pequeñas moléculas VE-822 y AZD-7762.[117] Estos sistemas permiten el control condicional de la actividad CRISPR para una mayor precisión, eficiencia y control espacio-temporal. El control espacio-temporal es una forma de eliminar los efectos fuera del objetivo: solo ciertas células o partes del organismo pueden necesitar ser modificadas, y por lo tanto, la luz o las pequeñas moléculas pueden usarse como una forma de llevar esto a cabo. La eficiencia del sistema CRISPR-Cas9 también aumenta significativamente mediante la entrega adecuada de las instrucciones de ADN para crear las proteínas y los reactivos necesarios.[117]
Las CRISPR también utilizan proteínas de edición de pares de bases individuales para crear ediciones específicas en una o dos bases en la secuencia objetivo. Las CRISPR/Cas9 se fusionaron con enzimas específicas que inicialmente solo podían cambiar mutaciones de C a T y de G a A y sus reversas. Esto se logró finalmente sin requerir ninguna escisión de ADN.[118][119][120] Con la fusión de otra enzima, el sistema de edición de bases CRISPR-Cas9 también puede editar de C a G y su reversa.[121]
Cribado CRISPR
editarEl sistema de repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas (CRISPR)/Cas9 es una tecnología de edición de genes que puede inducir roturas de doble cadena (DSBs, por sus siglas en inglés: double strand breaks) en cualquier lugar en el que los ácidos ribonucleicos guía (ARNg) puedan unirse a la secuencia del motivo adyacente al protoespaciador (PAM).[122] Las mellas de cadena sencilla también pueden ser inducidas por mutantes del sitio activo de Cas9,[123] también conocidos como mellasas Cas9.[124] Simplemente cambiando la secuencia del ARNg, la Cas9-endonucleasa puede llegar a un gen de interés e inducir DSBs.[125] La eficacia de la endonucleasa Cas9 y la facilidad con la que se puede atacar a los genes condujeron al desarrollo de las bibliotecas CRISPR-knockout (KO), tanto para células de ratón como humanas, que pueden abarcar conjuntos de genes específicos de interés o todo el genoma.[126][127] El cribado CRISPR ayuda a los científicos a crear una perturbación genética sistemática y de alto rendimiento en organismos modelo vivos. Esta perturbación genética es necesaria para comprender plenamente la función génica y la regulación epigenética.[128] La ventaja de las bibliotecas CRISPR combinadas es que pueden atacarse más genes a la vez.
Las bibliotecas de knockout se crean de manera que logren una representación y rendimiento iguales en todos los ARNg expresados, y llevan un marcador de selección de antibióticos o fluorescente que se puede utilizar para recuperar células transducidas.[122] Hay dos sistemas de plásmidos en las bibliotecas de CRISPR/Cas9. Primero, está el plásmido "todo en uno", donde ARNgu y Cas9 se producen simultáneamente en una célula transfecatada. Segundo, es un sistema de dos vectores: los plásmidos sgRNA y Cas9 se entregan por separado.[129] Es importante entregar miles de vectores únicos que contienen ARNgu a un solo recipiente de células mediante transducción viral a una baja multiplicidad de infección (MOI, típicamente de 0.1 a 0.6), ya que esto evita la probabilidad de que un clon celular individual reciba más de un tipo; de lo contrario, podría conducir a una asignación incorrecta del genotipo al fenotipo.[127]
Una vez preparada la biblioteca agrupada, es necesario llevar a cabo una secuenciación profunda (NGS, secuenciación de nueva generación del ADN plasmídico amplificado por PCR para revelar la abundancia de sgRNAs. Las células de interés pueden ser consecuentemente infectadas por la biblioteca y luego seleccionadas según el fenotipo. Hay 2 tipos de selección: negativa y positiva. Con la selección negativa, las células muertas o de crecimiento lento se detectan eficientemente. Esto puede identificar genes esenciales para la supervivencia, que pueden servir además como candidatos para fármacos dirigidos molecularmente. Por otro lado, la selección positiva proporciona una colección de poblaciones adquiridas con ventaja de crecimiento por mutagénesis aleatoria.[122] Después de la selección, se recoge el ADN genómico y se secuencia mediante NGS. Se detecta el agotamiento o enriquecimiento de ARNgu y se compara con la biblioteca original de ARNsg, anotada con el gen objetivo al que corresponde el ARNgu. El análisis estadístico luego identifica los genes que probablemente sean relevantes para el fenotipo de interés.[127]
| Biblioteca | ID | Especie | PI | Genes objetivo | ARNg por gen | ARNg total |
|---|---|---|---|---|---|---|
| Bassik Mouse CRISPR Knockout Library | 1000000121–1000000130 | Ratón | Bassik | Varía (~23,000 en total) | ~10 | Varía |
| Mouse Tumor Suppressor Gene CRISPR Knockout Library | 113584 EFS backbone
113585 TBG backbone |
Ratón | Chen | 56 | ~4 | 286 |
| Brie mouse genome-wide library | 73632 (1 plasmid)
73633 (2 plasmid) |
Ratón | Doench y Root | 19,674 | 4 | 78,637 |
| Bassik Human CRISPR Knockout Library | 101926–101934 | Humana | Bassik | Varía (~20,500 en total) | ~10 | Varía |
| Brunello human genome-wide library | Humana | Doench y Root | 19,114 | 4 | 76,441 | |
| Mini-human AsCpf1-based Human Genome-wide Knockout Library | 130630 | Humana | Draetta | 16,977 | 3–4 | 17,032 matrices |
Además de las bibliotecas de eliminación (knock-out), también existen bibliotecas de reducción (knock-down, CRISPRi) y de activación (activation, CRISPRa), las cuales utilizan la capacidad de las proteínas de fusión Cas9 inactivadas proteolíticamente (dCas9) para unirse al ADN objetivo, lo que significa que el gen de interés no se corta, sino que se sobreexpresa o se reprime. Esto hace que el sistema CRISPR/Cas9 sea aún más interesante en la edición génica. La proteína dCas9 inactiva modula la expresión génica al dirigir represores o activadores de dCas9 hacia los promotores o sitios de inicio de la transcripción de los genes objetivo. Para reprimir genes, Cas9 puede fusionarse con el dominio efector KRAB, que forma un complejo con ARNg, mientras que CRISPRa utiliza dCas9 fusionado con diferentes dominios de activación transcripcional, los cuales son dirigidos por ARNg hacia las regiones promotoras para aumentar la expresión.[131][132][133]
Aplicaciones
editarModelos de enfermedades
editarLa modificación genómica Cas9 ha permitido la generación rápida y eficiente de modelos transgénicos en el campo de la genética. El Cas9 puede introducirse fácilmente en las células objetivo junto con ARNgu a través de la transfección de plásmidos para modelar la propagación de enfermedades y la respuesta de las células y su defensa contra la infección.[134] La capacidad de Cas9 de ser introducido in vivo permite la creación de modelos más precisos de la función génica y los efectos de la mutación, evitando las mutaciones fuera del objetivo observadas típicamente con métodos más antiguos de ingeniería genética.
La revolución de las CRISPR y Cas9 en la modelación genómica no se limita solo a los mamíferos. Los modelos genómicos tradicionales como Drosophila melanogaster, uno de los primeros organismos modelo, han visto un mayor refinamiento en su resolución con el uso de Cas9.[134] El Cas9 utiliza promotores específicos de células que permiten un uso controlado del Cas9. El Cas9 es un método preciso para tratar enfermedades debido a la orientación de la enzima Cas9 que solo afecta a ciertos tipos de células. Las células sometidas a la terapia de Cas9 también pueden ser eliminadas y reintroducidas para proporcionar efectos amplificados de la terapia.[135]
Las CRISPR-Cas9 puede ser utilizadas para editar el ADN de organismos in vivo y para eliminar genes individuales o incluso cromosomas enteros de un organismo en cualquier punto de su desarrollo. Existen cromosomas que han sido eliminados con éxito in vivo utilizando técnicas CRISPR, e incluyen el cromosoma Y y el cromosoma X de ratones de laboratorio adultos y los cromosomas humanos 14 y 21, en líneas de células madre embrionarias y ratones aneuploides respectivamente. Este método podría ser útil para tratar trastornos genéticos causados por números anormales de cromosomas, como el síndrome de Down y los trastornos intersexuales.[136]
La edición exitosa del genoma in vivo utilizando CRISPR-Cas9 se ha demostrado en numerosos organismos modelo, incluidos Escherichia coli,[137] Saccharomyces cerevisiae,[138][139] Candida albicans, Methanosarcina acetivorans,[140][141] Caenorhabditis elegans,[142] Arabidopsis spp.,[143] Danio rerio[144] y Mus musculus.[145][146] Se han logrado éxitos en el estudio de la biología básica, en la creación de modelos de enfermedades[142][147] y en el tratamiento experimental de modelos de enfermedades.[148]
Se han planteado preocupaciones de que los efectos fuera del objetivo (edición de genes además de los previstos) puedan confundir los resultados de un experimento de edición génica CRISPR (es decir, el cambio fenotípico observado puede no deberse a la modificación del gen objetivo, sino a otro gen). Se han realizado modificaciones a CRISPR para minimizar la posibilidad de efectos fuera del objetivo. Los experimentos CRISPR ortogonales a menudo se recomiendan para confirmar los resultados de un experimento de edición génica.[149][150]
Las CRISPR simplifican la creación de organismos genéticamente modificados para la investigación que imitan enfermedades o muestran qué sucede cuando se reduce o muta un gen. Las CRISPR puede utilizarse a nivel germinal para crear organismos en los que el gen objetivo se cambie en todas partes (es decir, en todas las células/tejidos/órganos de un organismo multicelular), o puede utilizarse en células no germinales para crear cambios locales que solo afecten a ciertas poblaciones celulares dentro del organismo.[151][152][153]
Las CRISPR pueden ser utilizadas para crear modelos celulares humanos de enfermedades.[154] Por ejemplo, cuando se aplica a células madre pluripotentes humanas, las CRISPR se han utilizado para introducir mutaciones dirigidas en genes relevantes para la enfermedad renal poliquística (PKD) y la glomeruloesclerosis focal y segmentaria (GEFS).[155] Estas células madre pluripotentes modificadas con las CRISPR fueron posteriormente cultivadas en organoides renales humanos que mostraron fenotipos específicos de la enfermedad.[156] Los organoides renales de células madre con mutaciones en un gen vinculado a la GEFS desarrollaron defectos de unión entre podocitos, las células de filtración afectadas en esa enfermedad. Esto se atribuyó a la incapacidad de los podocitos para formar microvellosidades entre células adyacentes.[157] Es importante destacar que estos fenotipos de enfermedad estaban ausentes en organoides de control de identidad genética idéntica, pero sin las modificaciones de las CRISPR.[155]
Se tomó un enfoque similar para modelar el síndrome del QT largo en cardiomiocitos derivados de células madre pluripotentes.[158] Estos modelos celulares generados por las CRISPR, con controles isogénicos, proporcionan una nueva forma de estudiar enfermedades humanas y probar medicamentos.
Biomedicina
editarLa tecnología CRISPR-Cas se ha propuesto como un tratamiento para múltiples enfermedades humanas, especialmente aquellas con causa genética.[159] Su capacidad para modificar secuencias de ADN específicas la convierte en una herramienta con potencial para corregir mutaciones que causan enfermedades. Las primeras investigaciones en modelos animales sugieren que las terapias basadas en la tecnología de las CRISPR tienen el potencial de tratar una amplia gama de enfermedades, [160]incluidos el cáncer,[161] la progeria,[162] la beta-talasemia,[163][164][165] la enfermedad de células falciformes,[165][166] la hemofilia,[167] la fibrosis quística,[168] la distrofia muscular de Duchenne,[169] la enfermedad de Huntington,[170][171] la amiloidosis transtiretina[43] y enfermedades cardíacas.[172] Las CRISPR también se han utilizado para curar la malaria en mosquitos, lo que podría eliminar el vector y la enfermedad en los humanos.[173] Las CRISPR también puede ntener aplicaciones en la ingeniería de tejidos y la medicina regenerativa, como la creación de vasos sanguíneos humanos que carecen de expresión de proteínas de clase II del MHC, que a menudo causan rechazo de trasplantes.[174]
Además, los ensayos clínicos para curar la beta talasemia y la enfermedad de células falciformes en pacientes humanos utilizando la tecnología CRISPR-Cas9 han mostrado resultados prometedores.[175][176]
Sin embargo, aún existen algunas limitaciones en el uso de la tecnología en terapia génica: la relativamente alta frecuencia de efectos fuera del objetivo, el requisito de una secuencia PAM cerca del sitio objetivo, la apoptosis mediada por p53 por roturas de doble cadena inducidas por CRISPR y la toxicidad inmunogénica debido al sistema de entrega típicamente por virus.[177]
Amaurosis congénita de Leber
editarEl tratamiento de las CRISPR para la LCA10 (la variante más común de la amaurosis congénita de Leber, que es la principal causa de ceguera infantil hereditaria) modifica el gen defectuoso del fotorreceptor del paciente.
En marzo de 2020, el primer voluntario paciente en este estudio en EE. UU., patrocinado por Editas Medicine, recibió una dosis baja del tratamiento para probar su seguridad.
En junio de 2021, comenzó la inscripción para un grupo de cohortes adultas y pediátricas de dosis alta de 4 voluntarios pacientes cada una. Se espera que la dosificación de las nuevas cohortes se complete para julio de 2022.[178] En noviembre de 2022, Editas informó que el 20% de los pacientes tratados tuvieron mejoras significativas, pero también anunciaron que la población objetivo resultante era demasiado pequeña para respaldar un desarrollo independiente continuo.[179]
Cáncer
editarLas CRISPR también han encontrado muchas aplicaciones en el desarrollo de inmunoterapias basadas en células.[180] El primer ensayo clínico que involucra las CRISPR comenzó en 2016. Implicó tomar células inmunes de personas con cáncer de pulmón, usar las CRISPR para editar el gen PD-1 expresado, y luego administrar las células alteradas de vuelta a la misma persona. Otros 20 ensayos estaban en marcha o casi listos, principalmente en China, hasta 2017.[161]
En 2016, la Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) aprobó un ensayo clínico en el que las CRISPR se utilizarían para alterar las células T extraídas de personas con diferentes tipos de cáncer y luego administrar esas células T modificadas de vuelta a las mismas personas.[181]
En noviembre de 2020, en modelos animales de ratón, las CRISPR se utilizaron de manera efectiva para tratar el glioblastoma (un tumor cerebral de rápido crecimiento) y el cáncer de ovario metastásico, ya que son dos cánceres con algunos de los peores pronósticos en el mejor de los casos y generalmente se diagnostican durante sus etapas finales. Los tratamientos han dado como resultado la inhibición del crecimiento del tumor y un aumento de la supervivencia en un 80% para el cáncer de ovario metastásico y la apoptosis de las células tumorales, la inhibición del crecimiento del tumor en un 50% y la mejora de la supervivencia en un 30% para el glioblastoma.[182]
En octubre de 2021, CRISPR Therapeutics anunció los resultados de su ensayo de fase 1 en curso basado en células T alogénicas en EE. UU. Estas células se obtienen de donantes sanos y se editan para atacar las células cancerosas y evitar ser percibidas como una amenaza por el sistema inmunológico del receptor, y luego se multiplican en grandes lotes que pueden ser administrados a grandes números de receptores.[178]
En diciembre de 2022, una niña británica de 13 años que había sido diagnosticada con leucemia linfoblástica aguda de células T incurable fue curada por médicos del Hospital Great Ormond Street, en el primer uso documentado de edición génica terapéutica para este propósito, después de someterse a seis meses de tratamiento experimental, donde intentos anteriores de otros tratamientos fallaron. El procedimiento incluyó reprogramar una célula T sana para destruir las células T cancerosas para primero liberarla de la leucemia, y luego reconstruir su sistema inmunológico desde cero utilizando células inmunes saludables.[183] El equipo utilizó la edición BASE y había tratado previamente un caso de leucemia linfoblástica aguda en 2015 utilizando TALENs.[184]
Diabetes
editarLa diabetes tipo 1 es un trastorno endocrino que resulta de la falta de células beta pancreáticas para producir insulina, un compuesto vital en el transporte de azúcar en la sangre a las células para producir energía. Los investigadores han estado tratando de trasplantar células beta saludables. Las CRISPR se usa para editar las células con el fin de reducir la probabilidad de que el cuerpo del paciente rechace el trasplante.
El 17 de noviembre de 2021, CRISPR Therapeutics y ViaCyte anunciaron que la agencia médica canadiense había aprobado su solicitud para un ensayo clínico para VCTX210, una terapia con células madre editadas con CRISPR diseñada para tratar la diabetes tipo 1. Esto fue significativo porque fue la primera terapia editada genéticamente para la diabetes que se acercaba a las clínicas. Las mismas compañías también desarrollaron un tratamiento novedoso para la diabetes tipo 1 para producir insulina a través de un pequeño implante médico que utiliza millones de células pancreáticas derivadas de células madre editadas con CRISPR.[185]
En febrero de 2022, se llevó a cabo un ensayo de fase 1 en el que un paciente voluntario recibió tratamiento.[178][186]
VIH/SIDA
editarEl virus de la inmunodeficiencia humana o VIH es un virus que ataca el sistema inmunológico del cuerpo. Si bien existen tratamientos efectivos que pueden permitir que los pacientes vivan vidas saludables, el VIH es retroactivo, lo que significa que incrusta una versión inactiva de sí mismo en el genoma humano. Las CRISPR pueden ser utilizadas para eliminar selectivamente el virus del genoma mediante el diseño de ARN guía para dirigirse al genoma retroactivo del VIH. Un problema con este enfoque es que requiere la eliminación del genoma del VIH de casi todas las células, lo que puede ser difícil de lograr en la práctica.[178]
Los resultados iniciales en el tratamiento y cura del VIH han sido bastante exitosos. En 2021, 9 de 23 ratones humanizados tratados con una combinación de antirretrovirales y CRISPR/Cas-9 dieron como resultado que el virus se volviera indetectable, incluso después del período de rebote habitual. Ninguno de los dos tratamientos por separado tuvo tal efecto.[187] Los ensayos clínicos en humanos de una terapia basada en CRISPR–Cas9, EBT-101, comenzarán en 2022.[188][189] En octubre de 2023, un estudio en fase inicial en 3 personas de EBT-101 informó que el tratamiento parecía ser seguro sin efectos secundarios importantes, pero no se divulgaron datos sobre su efectividad.[190] En 2024, otra terapia CRISPR de investigadores de la Universidad de Ámsterdam informó de la eliminación del VIH en cultivos celulares.[191][192]
Infección
editarLas "nucleasas guiadas por ARN" basadas en CRISPR-Cas pueden usarse para apuntar a factores de virulencia, genes que codifican resistencia a antibióticos y otras secuencias de interés médico. Esta tecnología representa así una forma novedosa de terapia antimicrobiana y una estrategia para manipular poblaciones bacterianas.[193][194] Estudios recientes sugieren una correlación entre la interferencia del locus CRISPR-Cas y la adquisición de resistencia a antibióticos.[195] Este sistema proporciona protección a las bacterias contra el ADN extraño invasor, como transposones, bacteriófagos y plásmidos. Este sistema se mostró como una fuerte presión selectiva para la adquisición de resistencia a antibióticos y factores de virulencia en patógenos bacterianos.[195]
Las terapias basadas en la tecnología de edición de genes CRISPR–Cas3 entregadas por bacteriófagos modificados podrían usarse para destruir ADN específico en patógenos.[196] La Cas3 es más destructivo que el mejor conocido Cas9.[197][198]
La investigación sugiere que las CRISPR son una forma efectiva de limitar la replicación de varios herpesvirus. Pudo erradicar el ADN viral en el caso del virus de Epstein-Barr (VEB). Las CRISPRs antivirales del herpes tienen aplicaciones prometedoras, como eliminar el VEB que causa cáncer de las células tumorales, ayudar a eliminar los órganos donados de los pacientes inmunocomprometidos de invasores virales o prevenir los brotes de herpes labial y las infecciones oculares recurrentes al bloquear la reactivación del VHS-1. Hasta agosto de 2016, estos estaban esperando pruebas.[199][200]
Las CRISPR pueden revivir el concepto de trasplantar órganos de animales en personas. Los retrovirus presentes en los genomas animales podrían dañar a los receptores de trasplantes. En 2015, un equipo eliminó 62 copias de una secuencia de ADN retroviral particular del genoma del cerdo en una célula epitelial renal.[201] Los investigadores demostraron recientemente la capacidad de dar a luz ejemplares de cerdo vivos después de eliminar estos retrovirus de su genoma utilizando las CRISPR por primera vez.[202]
Enfermedades neurológicas
editarLas CRISPR son las únicas en el desarrollo para resolver enfermedades neurológicas por varias razones. Por ejemplo, las CRISPR permiten a los investigadores generar rápidamente modelos de células animales y humanas. Esto les permite estudiar cómo funcionan los genes en un sistema nervioso. Al introducir mutaciones que se relacionan con diversas enfermedades dentro de estas células, los investigadores pueden estudiar los efectos de los cambios en el desarrollo, la función y el comportamiento del sistema nervioso.[203] Pueden descubrir los mecanismos moleculares que contribuyen a estos trastornos, lo cual es esencial para desarrollar tratamientos efectivos. Esto es particularmente útil en la modelización y tratamiento de trastornos neurológicos complejos como el Alzheimer, Parkinson y la epilepsia, entre otros.
La enfermedad de Alzheimer (EA) es una enfermedad neurodegenerativa caracterizada por la pérdida de neuronas y la acumulación de ovillos neurofibrilares intracelulares y placas amiloides extracelulares en el cerebro.[204] Se han identificado tres genes patogénicos conocidos que causan el inicio temprano de la EA en humanos, específicamente la proteína precursora del amiloide (APP), la presenilina 1 (PSEN1) y la presenilina 2 (PSEN2).[204] Se han detectado más de 300 mutaciones en estos genes, lo que resulta en un aumento en el total de β-amiloide (Aβ), la relación Aβ42/40 y/o la polimerización de Aβ.
En el caso de la distrofia muscular de Duchenne, la mutación responsable de la enfermedad ocurre en el gen de la distrofina.[205] Las CRISPR se ha utilizado para corregir esto. Del mismo modo, para el síndrome de Dravet, que es un trastorno epiléptico, las CRISPR se han utilizado para corregir la mutación del gen SCN1A.[206] A pesar del progreso que se ha logrado, todavía existen desafíos en el uso de las CRISPR. Debido al hecho de que el cerebro está compuesto por una barrera hematoencefálica, es difícil transferir los componentes de las CRISPR a través de esta. Sin embargo, los avances recientes en sistemas de entrega de nanopartículas y vectores virales han mostrado expectativas para superar este obstáculo. En un futuro se espera que el uso de las CRISPR en neurociencia aumente a medida que la tecnología evoluciona.
Antropología genética
editarLas CRISPR-Cas9 se pueden utilizar para investigar e identificar las diferencias genéticas entre los humanos y otros simios, especialmente del cerebro. Por ejemplo, reintroduciendo variantes génicas arcaicas en organoides cerebrales para mostrar un impacto en la neurogénesis,[207] la longitud de la metafase de los progenitores apicales del neocórtex en desarrollo,[208] o mediante la eliminación de un gen en células madre embrionarias para identificar un regulador genético que, a través de la transición temprana de la forma celular, impulsa la expansión evolutiva del cerebro humano.[209][210] Un estudio describió un impacto importante de una variante génica arcaica en el neurodesarrollo[211][212] que puede ser un artefacto de un efecto secundario de las CRISPR,[213][214] ya que no pudo replicarse en un estudio posterior.[79]
Según la técnica
editarSupresión (knockdown)/activación
editarEl uso de versiones "muertas" de la Cas9 (dCas9) elimina la capacidad de corte de ADN de las CRISPR, mientras conserva su capacidad para dirigirse a secuencias deseables. Múltiples grupos han añadido diversos factores regulatorios a la dCas9, permitiéndoles encender o apagar casi cualquier gen o ajustar su nivel de actividad.[201] Al igual que el RNA de interferencia (RNAi), la interferencia de las CRISPR (CRISPRi) apaga los genes de manera reversible al dirigirse, pero no cortar un sitio. El sitio objetivo se metila, modificando epigenéticamente el gen. Esta modificación inhibe la transcripción. Estas modificaciones colocadas con precisión pueden luego utilizarse para regular los efectos en las expresiones génicas y la dinámica del ADN después de la inhibición de ciertas secuencias genómicas dentro del ADN. En los últimos años se han investigado de cerca las marcas epigenéticas en diferentes células humanas y se han encontrado ciertos patrones dentro de las marcas que se correlacionan con todo, desde el crecimiento tumoral hasta la actividad cerebral.[10] Por el contrario, la activación mediada por CRISPR (CRISPRa) promueve la transcripción génica.[215] La Cas9 es una forma eficaz de dirigir y silenciar genes específicos a nivel de ADN.[216] En las bacterias, la presencia de la Cas9 por sí sola es suficiente para bloquear la transcripción. Para aplicaciones en mamíferos, se añade una sección de proteína. Su ARN guía se dirige a secuencias de ADN regulatorias llamadas promotores que preceden inmediatamente al gen objetivo.[217]
La Cas9 se utilizó para transportar factores de transcripción sintéticos que activaban genes humanos específicos. La técnica logró un fuerte efecto al dirigir múltiples construcciones de CRISPR a ubicaciones ligeramente diferentes en el promotor del gen.[217]
Edición de ARN
editarEn 2016, los investigadores demostraron que las CRISPR de una bacteria bucal ordinaria podría usarse para editar ARN. Los investigadores buscaron en bases de datos que contenían cientos de millones de secuencias genéticas aquellas que se parecían a los genes CRISPR. Consideraron al Fusobacterium Leptotrichia shahii. Tenía un grupo de genes que se parecían a los genes CRISPR, pero con diferencias importantes. Cuando los investigadores equiparon a otras bacterias con estos genes, a los que llamaron C2c2, descubrieron que los organismos adquirieron una defensa novedosa.[218] C2c2 ha sido renombrado posteriormente como Cas13a para ajustarse a la nomenclatura estándar para los genes Cas.[219]
Muchos virus codifican su información genética en ARN en lugar de ADN, que reutilizan para hacer nuevos virus. El VIH y el poliovirus son tales virus. Las bacterias con Cas13 producen moléculas que pueden desmembrar el ARN, destruyendo el virus. Adaptar estos genes abre cualquier molécula de ARN para su edición.[218]
Los sistemas CRISPR-Cas también se pueden emplear para la edición de genes de micro-ARN y ARN no codificantes largos en plantas.[220]
Aplicaciones terapéuticas
editarDirigir ediciones para corregir secuencias mutadas fue propuesto y demostrado por primera vez en 1995.[221] Este trabajo inicial utilizó oligonucleótidos antisentido de ARN sintético complementarios a una mutación de codón de parada prematuro en una secuencia de distrofina para activar la edición A-a-I del codón de terminación a un codón de lectura a través en un sistema de células xenopus modelo.[221] Si bien esto también llevó a transiciones inadvertidas cercanas de A a I, las transiciones de A a I (leídas como G) pueden corregir los tres codones de terminación, pero no pueden crear uno. Por lo tanto, los cambios llevaron a una corrección de >25% del codón de terminación objetivo con lectura a través de una secuencia de reportero luciferasa. El trabajo posterior de Rosenthal logró la edición de la secuencia de ARNm mutada en cultivos celulares de mamíferos al dirigir un oligonucleótido unido a una desaminasa de citidina para corregir una secuencia de fibrosis quística mutada.[222] Más recientemente, se ha empleado CRISPR-Cas13 fusionado con desaminasas para dirigir la edición de ARNm.[223]
En 2022, se informó sobre la edición terapéutica de ARN para Cas7-11,[224][225] la cual permite cortes lo suficientemente dirigidos y una versión anterior de él se utilizó para la edición in vitro en 2021.[226]
Comparación con la edición de ADN
editarA diferencia de la edición de ADN, que es permanente, los efectos de la edición de ARN, incluidas las posibles mutaciones fuera del objetivo en el ARN, son transitorios y no se heredan. Por lo tanto, se considera que la edición de ARN es menos arriesgada. Además, puede requerir solo un ARN guía mediante el uso de la proteína ADAR ya presente en las células humanas y de muchos otros eucariotas, en lugar de necesitar introducir una proteína extranjera en el cuerpo.[227]
Genética dirigida
editarLa genética dirigida puede proporcionar una herramienta poderosa para restaurar el equilibrio de los ecosistemas mediante la eliminación de especies invasoras. Se han planteado preocupaciones sobre la eficacia y las consecuencias no deseadas en la especie objetivo, así como en las especies no objetivo, especialmente en la posibilidad de liberación accidental de laboratorios al medio ambiente. Los científicos han propuesto varias medidas de seguridad para garantizar la contención de los impulsos génicos experimentales, incluidas las medidas moleculares, reproductivas y ecológicas.[228] Muchos recomiendan que los impulsos de inmunización e inversión se desarrollen junto con los impulsos génicos para sobrescribir sus efectos si es necesario.[229] Existe consenso en que los efectos a largo plazo deben estudiarse más a fondo, especialmente en la posible interrupción ecológica que no se puede corregir con impulsos de reversión.[230]
Depleción genética in vitro
editarLas bibliotecas de secuenciación no enriquecidas a menudo tienen abundantes secuencias no deseadas. La Cas9 puede depletar específicamente las secuencias no deseadas con rotura de doble cadena con hasta un 99% de eficacia y sin efectos fuera del objetivo significativos como se ve con las enzimas de restricción. El tratamiento con Cas9 puede agotar el ARNr abundante mientras aumenta la sensibilidad a los patógenos en las bibliotecas de ARN-seq.[231]
Edición del epigenoma
editarLa edición del epigenoma o la ingeniería del epigenoma es un tipo de ingeniería genética en la que el epigenoma se modifica en sitios específicos utilizando moléculas diseñadas dirigidas a esos sitios (a diferencia de las modificaciones de todo el genoma). Mientras que la edición de genes implica cambiar la secuencia de ADN en sí, la edición epigenética implica modificar secuencias de ADN y otros factores de unión al ADN que influyen en su funcionamiento. Al "editar" características epigenómicas se puede determinar el papel biológico exacto de una modificación epigenética.
Las proteínas diseñadas que se utilizan para la edición del epigenoma están compuestas por un dominio de unión al ADN que se dirige a secuencias específicas y un dominio efector que modifica las características epigenómicas. Se han utilizado predominantemente tres grupos principales de proteínas de unión al ADN para la edición del epigenoma: proteínas con dedos de zinc, efectores similares a activadores de la transcripción (TALE) y fusiones de Cas9 deficientes en nucleasa (CRISPR).Aplicaciones
editarLa regulación dirigida de genes relacionados con enfermedades puede permitir nuevas terapias para muchas enfermedades, especialmente en los casos en los que aún no se han desarrollado terapias génicas adecuadas o son inapropiadas.[232] Si bien las consecuencias transgeneracionales y a nivel poblacional no se comprenden completamente, puede convertirse en una herramienta importante para la genómica funcional aplicada y la medicina personalizada.[233] Al igual que con la edición de ARN, no implica cambios genéticos ni los riesgos que los acompañan.[232] En 2021 se describió un ejemplo de un posible uso funcional de la edición del epigenoma: la represión de la expresión del gen Nav1.7 mediante CRISPR-dCas9, que mostró potencial terapéutico en tres modelos de ratón con dolor crónico.[234][235]
Una investigación evaluó su utilidad para reducir los niveles de proteína tau, regular una proteína implicada en la enfermedad de Huntington, combatir una forma hereditaria de obesidad y el síndrome de Dravet.[236]Integrasas dirigidas por CRISPR
editarLa combinación de CRISPR-Cas9 con integrasas permitió una técnica para ediciones grandes[Nota 1] sin problemas de roturas de doble cadena, como se demostró con PASTE[Nota 2] en 2022. Los investigadores informaron que podría usarse para entregar genes de hasta 36,000 pares de bases de ADN a varios tipos de células humanas y, por lo tanto, potencialmente para tratar enfermedades causadas por un gran número de mutaciones.[237][238]
Edición de calidad
editarLa edición de calidad[239] (o edición base) es un refinamiento de CRISPR para insertar o eliminar secciones de ADN con precisión. Las ediciones de CRISPR no siempre son perfectas y los cortes pueden terminar en el lugar incorrecto; ambos problemas son un problema para usar la tecnología en medicina.[240] La edición de calidad no corta el ADN de doble cadena, sino que utiliza el aparato de destino de las CRISPR para transportar una enzima adicional a una secuencia deseada, donde convierte un nucleótido único en otro.[241] La nueva guía, llamada pegRNA, contiene una plantilla de ARN para una nueva secuencia de ADN que se agregará al genoma en el lugar de destino. Eso requiere una segunda proteína, unida a la Cas9: una enzima transcriptasa inversa, que puede hacer una nueva cadena de ADN a partir de la plantilla de ARN e insertarla en el sitio marcado.[242] Esos tres eventos de emparejamiento independientes brindan una oportunidad para evitar secuencias fuera del objetivo, lo que aumenta significativamente la flexibilidad del objetivo y la precisión de la edición.[241] La edición de calidad fue desarrollada por investigadores del Instituto Broad del MIT y Harvard en Massachusetts.[243] Se necesita más investigaciones para optimizar estos métodos.[242][243]
Sociedad y cultura
editarModificación de la línea germinal humana
editarHasta marzo de 2015, varios grupos habían anunciado investigaciones en curso con la intención de sentar las bases para aplicar las CRISPR a embriones humanos para la ingeniería de la línea germinal humana, incluidos laboratorios en EE. UU., China y el Reino Unido, así como la empresa biotecnológica estadounidense OvaScience.[244] Científicos, incluido un co-descubridor de las CRISPR, instaron a una moratoria mundial sobre la aplicación de las CRISPR a la línea germinal humana, especialmente para uso clínico. Ellos explicaron que "los científicos deberían evitar incluso intentar, en jurisdicciones laxas, la modificación del genoma de la línea germinal para aplicación clínica en humanos" hasta que las implicaciones completas "se discutan entre organizaciones científicas y gubernamentales".[245][246] Estos científicos apoyan una investigación de bajo nivel adicional sobre las CRISPR y no consideran que las CRISPR estén lo suficientemente desarrolladas para ningún uso clínico en la realización de cambios heredables en humanos.[247]
En abril de 2015, científicos chinos informaron los resultados de un intento de alterar el ADN de embriones humanos no viables usando las CRISPR para corregir una mutación que causa talasemia beta, un trastorno hereditario letal.[248][249] El estudio había sido rechazado previamente tanto por Nature como por Science, en parte debido a preocupaciones éticas.[250] Los experimentos resultaron en el cambio exitoso de solo algunos de los genes previstos y tuvieron efectos fuera del objetivo en otros genes. Los investigadores declararon que CRISPR no está listo para su aplicación clínica en medicina reproductiva.[250] En abril de 2016, se informó que los científicos chinos habían realizado un segundo intento infructuoso de alterar el ADN de embriones humanos no viables utilizando las CRISPR, esta vez para alterar el gen CCR5 para hacer que el embrión fuera resistente a la infección por VIH.[251]
En diciembre de 2015, tuvo lugar una Cumbre Internacional sobre Edición del Genoma Humano en Washington bajo la dirección de David Baltimore. Los miembros de las academias científicas nacionales de EE. UU., Reino Unido y China discutieron la ética de la modificación de la línea germinal. Acordaron apoyar la investigación básica y clínica bajo ciertas pautas legales y éticas. Se hizo una distinción específica entre células somáticas, donde los efectos de las ediciones se limitan a un solo individuo, y células germinales, donde los cambios en el genoma pueden ser heredados por los descendientes. Las modificaciones heredables podrían tener consecuencias no deseadas y de largo alcance para la evolución humana, genéticamente (por ejemplo, interacciones gen-ambiente) y culturalmente (por ejemplo, darwinismo social). La alteración de gametocitos y embriones para generar cambios heredables en los humanos se definió como irresponsable. El grupo acordó iniciar un foro internacional para abordar tales preocupaciones y armonizar regulaciones en todos los países.[252]
En febrero de 2017, el Comité de Edición del Genoma Humano de las Academias Nacionales de Ciencias, Ingeniería y Medicina de los Estados Unidos (NASEM) publicó un informe que revisaba preocupaciones éticas, legales y científicas sobre la tecnología de ingeniería genómica. La conclusión del informe afirmaba que la edición genómica heredable es inadmisible en este momento, pero podría justificarse para ciertas condiciones médicas; sin embargo, no justificaron el uso de las CRISPR para mejoras.[253]
En noviembre de 2018, Jiankui He anunció que había editado dos embriones humanos para intentar desactivar el gen CCR5, que codifica un receptor que el VIH utiliza para ingresar a las células. Dijo que las gemelas, Lulu y Nana, habían nacido unas semanas antes. Dijo que las niñas todavía llevaban copias funcionales de CCR5 junto con CCR5 desactivado (mosaicismo) y todavía eran vulnerables al VIH. El trabajo fue ampliamente condenado como poco ético, peligroso y prematuro.[254] Un grupo internacional de científicos pidió una moratoria global sobre la edición genética de embriones humanos.[255]
Creación de bebés "a medida"
editarLa llegada de la tecnología de edición genética CRISPR-Cas9 ha llevado a la posibilidad de crear "bebés a medida". Esta tecnología tiene la posibilidad de eliminar ciertas enfermedades genéticas o mejorar la salud mediante el fortalecimiento de ciertos rasgos genéticos.
Barreras políticas para la ingeniería genética
editarLas regulaciones políticas para el sistema CRISPR-Cas9 varían en todo el mundo. En febrero de 2016, a los científicos británicos se les otorgó permiso de los reguladores para modificar genéticamente embriones humanos utilizando CRISPR-Cas9 y técnicas relacionadas. Sin embargo, se les prohibió a los investigadores implantar los embriones y los embriones debían ser destruidos después de siete días.[256]
En los Estados Unidos, existe un sistema regulador interdepartamental elaborado para evaluar nuevos alimentos y cultivos modificados genéticamente. Por ejemplo, la Ley de Protección de Riesgos Agrícolas de 2000 otorga al Departamento de Agricultura de los Estados Unidos (o por sus siglas en inglés, USDA) la autoridad para supervisar la detección, control, erradicación, supresión, prevención o retardo de la propagación de plagas vegetales o malezas nocivas para proteger la agricultura, el medio ambiente y la economía de los EE. UU. La ley regula cualquier organismo modificado genéticamente que utilice el genoma de una "plaga vegetal" predefinida o cualquier planta no previamente categorizada.[257] En 2015, Yinong Yang desactivó con éxito 16 genes específicos en el champiñón blanco para evitar que se oscurecieran. Dado que no había agregado ADN de especies extranjeras (transgénico) a su organismo, el champiñón no podía ser regulado por el USDA según la Sección 340.2.[258] El champiñón blanco de Yang fue el primer organismo modificado genéticamente con el sistema de proteínas CRISPR-Cas9 que pasó la regulación estadounidense.[259]
En 2016, el USDA patrocinó un comité para considerar la futura política regulatoria para las próximas técnicas de modificación genética. Con la ayuda de las Academias Nacionales de Ciencias, Ingeniería y Medicina de los Estados Unidos, grupos de intereses especiales se reunieron el 15 de abril para contemplar los posibles avances en la ingeniería genética en los próximos cinco años y cualquier nueva regulación que pudiera ser necesaria como resultado.[260] En 2017, la Administración de Alimentos y Medicamentos propuso una norma que clasificaría las modificaciones de ingeniería genética en animales como "medicamentos para animales", sometiéndolos a una regulación estricta si se ofrecieran a la venta y reduciendo la capacidad para que individuos y pequeñas empresas los hicieran rentables.[261][262]
En China, donde las condiciones sociales contrastan fuertemente con las del Occidente, las enfermedades genéticas llevan un estigma importante.[263] Esto deja a China con menos barreras políticas para el uso de esta tecnología.[264][265]
Reconocimiento
editarEn 2012 y 2013, las CRISPR fueron finalistas en el premio al Breakthrough of the Year de la revista Science. En 2015, fue el ganador de ese premio.[201] Las CRISPR fueron nombradas como una de las 10 tecnologías innovadoras por la MIT Technology Review en 2014 y 2016.[266][267] En 2016, Jennifer Doudna y Emmanuelle Charpentier, junto con Rudolph Barrangou, Philippe Horvath y Feng Zhang, ganaron el premio internacional Gairdner. En 2017, Doudna y Charpentier recibieron el Premio Japón en Tokio, Japón, por su revolucionaria invención de CRISPR-Cas9. En 2016, Charpentier, Doudna y Zhang ganaron el Premio Tang en Ciencias Biofarmacológicas.[268] En 2020, Charpentier y Doudna recibieron el Premio Nobel de Química, el primero de su tipo para un equipo completamente femenino, "por el desarrollo de un método para la edición del genoma".[269]
Véase también
editarNotas
editarReferencias
editar- ↑ a b c Bak, Rasmus O.; Gomez-Ospina, Natalia; Porteus, Matthew H. (2018-08). «Gene Editing on Center Stage». Trends in Genetics 34 (8): 600-611. ISSN 0168-9525. doi:10.1016/j.tig.2018.05.004. Consultado el 31 de mayo de 2024.
- ↑ «The Nobel Prize in Chemistry 2020». NobelPrize.org (en inglés estadounidense). Consultado el 31 de mayo de 2024.
- ↑ Cohen, J. (7 de octubre de 2020). «CRISPR, the revolutionary genetic ‘scissors,' honored by Chemistry Nobel». Science (en inglés). doi:10.1126/science.abf0540. Consultado el 31 de mayo de 2024.
- ↑ Cohen J. (4 de junio de 2018). «With prestigious prize, an overshadowed CRISPR researcher wins the spotlight». Science | AAAS (en inglés).
- ↑ a b Owens, Rebecca (8 de octubre de 2020). «Nobel prize: who gets left out?». The Conversation (en inglés estadounidense). Consultado el 31 de mayo de 2024.
- ↑ «Lithuanian scientists not awarded Nobel prize despite discovering same technology». lrt.lt (en inglés). 8 de octubre de 2020. Consultado el 31 de mayo de 2024.
- ↑ Šikšnys V. «Imam genų žirkles, iškerpam klaidą, ligos nelieka» (en lituano). Laisvės TV / Freedom TV. LaisvėsTV. min. 12:22. https://www.youtube.com/watch?v=_NQqKMyXSYQ&t=12m22s. «<...>Tai mes tą savo straipsnį išsiuntėm į redakciją pirmieji, bet laimės ten daug nebuvo. Viena redakcija pasakė, kad mes net recenzentam nesiųsim. Nusiuntėm į kitą redakciją – tai jis (straipsnis) pragulėjo kažkur ant redaktoriaus stalo labai ilgai. Na ir taip galų gale išsiuntėm į trečią žurnalą ir trečias žurnalas po kelių mėnesių jį išspausdino. Bet, aišku, Berklio universiteto mokslininkams sekėsi geriau – jie išsiuntė straipsnį į žurnalą Science – jį priėmė ir išspausdino per 2 savaites. Nors iš tikro jie tą straispnį išsiuntė pora mėnesių vėliau nei mes. <...> Bueno, fuimos quienes enviaron el artículo primero, pero no tuvimos tanta suerte.».
- ↑ Zhang, Jian-Hua; Pandey, Mritunjay; Kahler, John F.; Loshakov, Anna; Harris, Benjamin; Dagur, Pradeep K.; Mo, Yin-Yuan; Simonds, William F. (2014-11). «Improving the specificity and efficacy of CRISPR/CAS9 and gRNA through target specific DNA reporter». Journal of Biotechnology 189: 1-8. ISSN 0168-1656. PMC 4252756. PMID 25193712. doi:10.1016/j.jbiotec.2014.08.033. Consultado el 31 de mayo de 2024.
- ↑ Vakulskas, Christopher A.; Dever, Daniel P.; Rettig, Garrett R.; Turk, Rolf; Jacobi, Ashley M.; Collingwood, Michael A.; Bode, Nicole M.; McNeill, Matthew S. et al. (2018-08). «A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells». Nature Medicine (en inglés) 24 (8): 1216-1224. ISSN 1546-170X. PMC 6107069. PMID 30082871. doi:10.1038/s41591-018-0137-0. Consultado el 31 de mayo de 2024.
- ↑ a b c Ledford, Heidi (1 de marzo de 2016). «CRISPR: gene editing is just the beginning». Nature (en inglés) 531 (7593): 156-159. ISSN 1476-4687. doi:10.1038/531156a. Consultado el 31 de mayo de 2024.
- ↑ Travis, J. (17 de diciembre de 2015). «Breakthrough of the Year: CRISPR makes the cut». Science Magazine (en inglés). American Association for the Advancement of Science.
- ↑ Ledford, Heidi (1 de junio de 2015). «CRISPR, the disruptor». Nature (en inglés) 522 (7554): 20-24. ISSN 1476-4687. doi:10.1038/522020a. Consultado el 31 de mayo de 2024.
- ↑ a b «Casgevy: UK approves gene-editing drug for sickle cell». BBC News (en inglés británico). 16 de noviembre de 2023. Consultado el 31 de mayo de 2024.
- ↑ a b «MHRA authorises world-first gene therapy that aims to cure sickle-cell disease and transfusion-dependent β-thalassemia». GOV.UK (en inglés). Consultado el 31 de mayo de 2024.
- ↑ «FDA Approves First Gene Therapies to Treat Patients with Sickle Cell Disease». Food and Drug Administration (en inglés). Fri, 12/08/2023 - 11:10. Consultado el 31 de mayo de 2024.
- ↑ Young, S (11 de febrero de 2014). «CRISPR and Other Genome Editing Tools Boost Medical Research and Gene Therapy's Reach». MIT Technology Review (en inglés).
- ↑ a b Heidenreich, Matthias; Zhang, Feng (2016-01). «Applications of CRISPR–Cas systems in neuroscience». Nature Reviews Neuroscience (en inglés) 17 (1): 36-44. ISSN 1471-0048. PMC 4899966. PMID 26656253. doi:10.1038/nrn.2015.2. Consultado el 31 de mayo de 2024.
- ↑ Barrangou, Rodolphe; Doudna, Jennifer A. (2016-09). «Applications of CRISPR technologies in research and beyond». Nature Biotechnology (en inglés) 34 (9): 933-941. ISSN 1546-1696. doi:10.1038/nbt.3659. Consultado el 31 de mayo de 2024.
- ↑ Cox, David Benjamin Turitz; Platt, Randall Jeffrey; Zhang, Feng (2015-02). «Therapeutic genome editing: prospects and challenges». Nature Medicine (en inglés) 21 (2): 121-131. ISSN 1546-170X. doi:10.1038/nm.3793. Consultado el 31 de mayo de 2024.
- ↑ a b c d «CRISPR Timeline». Broad Institute (en inglés). 25 de septiembre de 2015. Consultado el 31 de mayo de 2024.
- ↑ «CRISPR Madness». GEN - Genetic Engineering and Biotechnology News (en inglés estadounidense). 8 de noviembre de 2013. Consultado el 31 de mayo de 2024.
- ↑ «Products & Services». Genetic Engineering & Biotechnology News (en inglés) 35 (21): 8-8. 2015-12. ISSN 1935-472X. doi:10.1089/gen.35.21.05. Consultado el 31 de mayo de 2024.
- ↑ «Who Owns the Biggest Biotech Discovery of the Century? There's a bitter fight over the patents for CRISPR, a breakthrough new form of DNA editing». MIT Technology Review (en inglés). Consultado el 31 de mayo de 2024.
- ↑ Fye, S. «Genetic Rough Draft: Editas and CRISPR». The Atlas Business Journal. Consultado el 31 de mayo de 2024.
- ↑ «CRISPR-Cas systems and methods for altering expression of gene products». Google Patents (en inglés).
- ↑ Shaffer, Catherine (14 de marzo de 2022). «Broad defeats Berkeley CRISPR patent». Nature Biotechnology (en inglés) 40 (4): 445-445. doi:10.1038/d41587-022-00004-2. Consultado el 31 de mayo de 2024.
- ↑ «CRISPR patents to go on trial». Nature Biotechnology (en inglés) 34 (2): 121-121. 1 de febrero de 2016. ISSN 1546-1696. doi:10.1038/nbt0216-121a. Consultado el 31 de mayo de 2024.
- ↑ Pollack, Andrew (16 de febrero de 2017). «Harvard and M.I.T. Scientists Win Gene-Editing Patent Fight». The New York Times (en inglés estadounidense). ISSN 0362-4331. Consultado el 31 de mayo de 2024.
- ↑ «Broad Wins CRISPR Patent Interference Case». The Scientist Magazine® (en inglés). Consultado el 31 de mayo de 2024.
- ↑ Noonan, K. E. (16 de febrero de 2017). «PTAB Decides CRISPR Interference in Favor of Broad Institute – Their Reasoning». Patent Docs (en inglés).
- ↑ Potenza, Alessandra (13 de abril de 2017). «UC Berkeley challenges decision that CRISPR patents belong to Broad Institute». The Verge (en inglés). Consultado el 31 de mayo de 2024.
- ↑ Buhr, Sarah (26 de julio de 2017). «The CRISPR patent battle is back on as UC Berkeley files an appeal». TechCrunch (en inglés estadounidense). Consultado el 31 de mayo de 2024.
- ↑ Wetsman, Nicole (1 de marzo de 2022). «UC Berkeley loses CRISPR patent case». The Verge (en inglés). Consultado el 31 de mayo de 2024.
- ↑ a b Philippidis, A. (7 de agosto de 2017). «MilliporeSigma to Be Granted European Patent for CRISPR Technology». GEN - Genetic Engineering and Biotechnology News (en inglés estadounidense). Consultado el 31 de mayo de 2024.
- ↑ «UC Berkeley Receives CRISPR Patent in Europe». The Scientist Magazine® (en inglés). Consultado el 31 de mayo de 2024.
- ↑ Cohen, Jon (4 de agosto de 2017). «CRISPR patent battle in Europe takes a ‘wild’ twist with surprising player». Science. ISSN 0036-8075. doi:10.1126/science.aan7211. Consultado el 31 de mayo de 2024.
- ↑ «Top EU court: GMO rules cover plant gene editing technique». Retuers (en inglés). 25 de julio de 2018.
- ↑ AFP (7 de febrero de 2020). «US Trial Shows 3 Cancer Patients Had Their Genomes Altered Safely by CRISPR». ScienceAlert (en inglés estadounidense). Consultado el 2 de junio de 2024.
- ↑ Chamary, J. V. «These Scientists Deserved A Nobel Prize, But Didn’t Discover Crispr». Forbes (en inglés). Consultado el 2 de junio de 2024.
- ↑ Fischman, Josh. «Nobel Prize in Chemistry Goes to Discovery of 'Genetic Scissors' Called CRISPR/Cas9». Scientific American (en inglés). Consultado el 2 de junio de 2024.
- ↑ «Two women share chemistry Nobel in historic win for 'genetic scissors'» (en inglés británico). 7 de octubre de 2020. Consultado el 2 de junio de 2024.
- ↑ Kaiser, J. (26 de junio de 2021). «CRISPR injected into the blood treats a genetic disease for first time». Science | AAAS (en inglés).
- ↑ a b Gillmore, Julian D.; Gane, Ed; Taubel, Jorg; Kao, Justin; Fontana, Marianna; Maitland, Michael L.; Seitzer, Jessica; O’Connell, Daniel et al. (5 de agosto de 2021). «CRISPR-Cas9 In Vivo Gene Editing for Transthyretin Amyloidosis». New England Journal of Medicine (en inglés) 385 (6): 493-502. ISSN 0028-4793. doi:10.1056/NEJMoa2107454. Consultado el 2 de junio de 2024.
- ↑ «Tomato In Japan Is First CRISPR-Edited Food In The World To Go On Sale». IFLScience (en inglés). 30 de septiembre de 2021. Consultado el 2 de junio de 2024.
- ↑ Boonstra, Evert; de Kleijn, Roy; Colzato, Lorenza S.; Alkemade, Anneke; Forstmann, Birte U.; Nieuwenhuis, Sander (6 de octubre de 2015). «Neurotransmitters as food supplements: the effects of GABA on brain and behavior». Frontiers in Psychology (en inglés) 6. ISSN 1664-1078. PMC 4594160. PMID 26500584. doi:10.3389/fpsyg.2015.01520. Consultado el 2 de junio de 2024.
- ↑ Wang, Tian; Zhang, Hongyan; Zhu, Hongliang (1 de diciembre de 2019). «CRISPR technology is revolutionizing the improvement of tomato and other fruit crops». Horticulture Research 6: 77. doi:10.1038/s41438-019-0159-x. Consultado el 2 de junio de 2024.
- ↑ «Japan embraces CRISPR-edited fish». Nature Biotechnology (en inglés) 40 (1): 10-10. 1 de enero de 2022. ISSN 1546-1696. doi:10.1038/s41587-021-01197-8. Consultado el 2 de junio de 2024.
- ↑ «Startup hopes genome-edited pufferfish will be a hit in 2022». The Japan Times (en inglés). 5 de enero de 2022. Archivado desde el original el 17 de enero de 2022.
- ↑ «Gene-edited sea bream set for sale in Japan». The Fish Site (en inglés). 22 de septiembre de 2021. Consultado el 2 de junio de 2024.
- ↑ Götz, Linde; Svanidze, Miranda; Tissier, Alain; Brand Duran, Alejandro (2022-01). «Consumers’ Willingness to Buy CRISPR Gene-Edited Tomatoes: Evidence from a Choice Experiment Case Study in Germany». Sustainability (en inglés) 14 (2): 971. ISSN 2071-1050. doi:10.3390/su14020971. Consultado el 2 de junio de 2024.
- ↑ «Are Consumers Willing to Buy CRISPR Tomatoes?». Crop Biotech Update (en inglés). Consultado el 2 de junio de 2024.
- ↑ «ゲノム編集技術を利用して開発した「可食部増量マダイ」、厚生労働省及び農林水産省への届出完了». プレスリリース・ニュースリリース配信シェアNo.1|PR TIMES (en jp). 17 de septiembre de 2021. Consultado el 2 de junio de 2024.
- ↑ Sanders, Robert (27 de mayo de 2021). «UC Berkeley will auction NFTs of Nobel Prize-winning inventions to fund research». Berkeley News (en inglés). Consultado el 2 de junio de 2024.
- ↑ Sestino, Andrea; Guido, Gianluigi; Peluso, Alessandro M. (2022). Non-Fungible Tokens (NFTs): Examining the Impact on Consumers and Marketing Strategies (en inglés). Springer International Publishing. ISBN 978-3-031-07202-4. doi:10.1007/978-3-031-07203-1. Consultado el 2 de junio de 2024.
- ↑ Chang, Kenneth (27 de mayo de 2021). «You Can Buy a Piece of a Nobel Prize-Winning Discovery». The New York Times (en inglés estadounidense). ISSN 0362-4331. Consultado el 2 de junio de 2024.
- ↑ Trautman, Lawrence J. (2021). «Virtual Art and Non-fungible Tokens». SSRN Electronic Journal (en inglés). ISSN 1556-5068. doi:10.2139/ssrn.3814087. Consultado el 2 de junio de 2024.
- ↑ Jones, Nicola (18 de junio de 2021). «How scientists are embracing NFTs». Nature (en inglés) 594 (7864): 481-482. doi:10.1038/d41586-021-01642-3. Consultado el 2 de junio de 2024.
- ↑ «FDA Approves First Gene Therapies to Treat Patients with Sickle Cell Disease». FDA (en inglés). Fri, 12/08/2023 - 11:10. Consultado el 2 de junio de 2024.
- ↑ a b c d e Ran, F. Ann; Hsu, Patrick D.; Wright, Jason; Agarwala, Vineeta; Scott, David A.; Zhang, Feng (2013-11). «Genome engineering using the CRISPR-Cas9 system». Nature Protocols (en inglés) 8 (11): 2281-2308. ISSN 1750-2799. PMC 3969860. PMID 24157548. doi:10.1038/nprot.2013.143. Consultado el 3 de junio de 2024.
- ↑ Ly, J. (2013). Discovering Genes Responsible for Kidney Diseases (Tesis de Ph.D.) (en inglés).
- ↑ Mohr, Stephanie E.; Hu, Yanhui; Ewen‐Campen, Benjamin; Housden, Benjamin E.; Viswanatha, Raghuvir; Perrimon, Norbert (2016-09). «CRISPR guide RNA design for research applications». The FEBS Journal (en inglés) 283 (17): 3232-3238. ISSN 1742-464X. PMC 5014588. PMID 27276584. doi:10.1111/febs.13777. Consultado el 3 de junio de 2024.
- ↑ Brazelton, Vincent A; Zarecor, Scott; Wright, David A; Wang, Yuan; Liu, Jie; Chen, Keting; Yang, Bing; Lawrence-Dill, Carolyn J (2 de octubre de 2015). «A quick guide to CRISPR sgRNA design tools». GM Crops & Food (en inglés) 6 (4): 266-276. ISSN 2164-5698. PMC 5033207. PMID 26745836. doi:10.1080/21645698.2015.1137690. Consultado el 3 de junio de 2024.
- ↑ «Researchers establish new viable CRISPR-Cas12b system for plant genome engineering». phys.org (en inglés). Consultado el 3 de junio de 2024.
- ↑ Ming, Meiling; Ren, Qiurong; Pan, Changtian; He, Yao; Zhang, Yingxiao; Liu, Shishi; Zhong, Zhaohui; Wang, Jiaheng et al. (2020-03). «CRISPR–Cas12b enables efficient plant genome engineering». Nature Plants (en inglés) 6 (3): 202-208. ISSN 2055-0278. doi:10.1038/s41477-020-0614-6. Consultado el 3 de junio de 2024.
- ↑ Cox, David B. T.; Gootenberg, Jonathan S.; Abudayyeh, Omar O.; Franklin, Brian; Kellner, Max J.; Joung, Julia; Zhang, Feng (24 de noviembre de 2017). «RNA editing with CRISPR-Cas13». Science (en inglés) 358 (6366): 1019-1027. ISSN 0036-8075. PMC 5793859. PMID 29070703. doi:10.1126/science.aaq0180. Consultado el 3 de junio de 2024.
- ↑ «CRISPR-Cas3 innovation holds promise for disease cures, advancing science | Cornell Chronicle». Cornell Chronicle (en inglés). Consultado el 3 de junio de 2024.
- ↑ Dolan, Adam E.; Hou, Zhonggang; Xiao, Yibei; Gramelspacher, Max J.; Heo, Jaewon; Howden, Sara E.; Freddolino, Peter L.; Ke, Ailong et al. (2019-06). «Introducing a Spectrum of Long-Range Genomic Deletions in Human Embryonic Stem Cells Using Type I CRISPR-Cas». Molecular Cell 74 (5): 936-950.e5. ISSN 1097-2765. doi:10.1016/j.molcel.2019.03.014. Consultado el 3 de junio de 2024.
- ↑ Liu, Zhenquan; Dong, Huina; Cui, Yali; Cong, Lina; Zhang, Dawei (3 de septiembre de 2020). «Application of different types of CRISPR/Cas-based systems in bacteria». Microbial Cell Factories 19 (1): 172. ISSN 1475-2859. doi:10.1186/s12934-020-01431-z. Consultado el 3 de junio de 2024.
- ↑ «Researchers develop an engineered 'mini' CRISPR genome editing system». phys.org (en inglés). Consultado el 3 de junio de 2024.
- ↑ Xu, Xiaoshu; Chemparathy, Augustine; Zeng, Leiping; Kempton, Hannah R.; Shang, Stephen; Nakamura, Muneaki; Qi, Lei S. (2021-10). «Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing». Molecular Cell 81 (20): 4333-4345.e4. ISSN 1097-2765. doi:10.1016/j.molcel.2021.08.008. Consultado el 3 de junio de 2024.
- ↑ Bravo, Jack P. K.; Liu, Mu-Sen; Hibshman, Grace N.; Dangerfield, Tyler L.; Jung, Kyungseok; McCool, Ryan S.; Johnson, Kenneth A.; Taylor, David W. (2022-03). «Structural basis for mismatch surveillance by CRISPR–Cas9». Nature (en inglés) 603 (7900): 343-347. ISSN 1476-4687. doi:10.1038/s41586-022-04470-1. Consultado el 3 de junio de 2024.
- ↑ «Protein tweak makes CRISPR gene editing 4,000 times less error-prone». New Atlas (en inglés estadounidense). 4 de marzo de 2022. Consultado el 3 de junio de 2024.
- ↑ Kato, Kazuki; Zhou, Wenyuan; Okazaki, Sae; Isayama, Yukari; Nishizawa, Tomohiro; Gootenberg, Jonathan S.; Abudayyeh, Omar O.; Nishimasu, Hiroshi (2022-06). «Structure and engineering of the type III-E CRISPR-Cas7-11 effector complex». Resumen por William S. . "Neuroscientists expand CRISPR toolkit with new, compact Cas7-11 enzyme", Massachusetts Institute of Technology.. Cell 185 (13): 2324-2337.e16. ISSN 0092-8674. doi:10.1016/j.cell.2022.05.003. Consultado el 3 de junio de 2024.
- ↑
- ↑ Roy, Sitara; Juste, Sara Sanz; Sneider, Marketta; Auradkar, Ankush; Klanseck, Carissa; Li, Zhiqian; Julio, Alison Henrique Ferreira; del Amo, Victor Lopez et al. (2022-07). «Cas9/Nickase-induced allelic conversion by homologous chromosome-templated repair in Drosophila somatic cells». Science Advances (en inglés) 8 (26). ISSN 2375-2548. doi:10.1126/sciadv.abo0721. Consultado el 4 de junio de 2024.
- ↑ Horvath, Philippe; Barrangou, Rodolphe (8 de enero de 2010). «CRISPR/Cas, the Immune System of Bacteria and Archaea». Science (en inglés) 327 (5962): 167-170. ISSN 0036-8075. doi:10.1126/science.1179555. Consultado el 4 de junio de 2024.
- ↑ Bialk, Pawel; Rivera-Torres, Natalia; Strouse, Bryan; Kmiec, Eric B. (8 de junio de 2015). «Regulation of Gene Editing Activity Directed by Single-Stranded Oligonucleotides and CRISPR/Cas9 Systems». PLOS ONE (en inglés) 10 (6): e0129308. ISSN 1932-6203. doi:10.1371/journal.pone.0129308. Consultado el 4 de junio de 2024.
- ↑ Sander, Jeffry D.; Joung, J. Keith (2014-04). «CRISPR-Cas systems for editing, regulating and targeting genomes». Nature Biotechnology (en inglés) 32 (4): 347-355. ISSN 1546-1696. PMC 4022601. PMID 24584096. doi:10.1038/nbt.2842. Consultado el 4 de junio de 2024.
- ↑ a b Riesenberg, Stephan; Kanis, Philipp; Macak, Dominik; Wollny, Damian; Düsterhöft, Dorothee; Kowalewski, Johannes; Helmbrecht, Nelly; Maricic, Tomislav et al. (2023-09). «Efficient high-precision homology-directed repair-dependent genome editing by HDRobust». Nature Methods (en inglés) 20 (9): 1388-1399. ISSN 1548-7105. PMC 10482697. PMID 37474806. doi:10.1038/s41592-023-01949-1. Consultado el 4 de junio de 2024.
- ↑ Lino, Christopher A.; Harper, Jason C.; Carney, James P.; Timlin, Jerilyn A. (1 de enero de 2018). «Delivering CRISPR: a review of the challenges and approaches». Drug Delivery (en inglés) 25 (1): 1234-1257. ISSN 1071-7544. doi:10.1080/10717544.2018.1474964. Consultado el 4 de junio de 2024.
- ↑ Li, Ling; Hu, Shuo; Chen, Xiaoyuan (2018-07). «Non-viral delivery systems for CRISPR/Cas9-based genome editing: Challenges and opportunities». Biomaterials 171: 207-218. ISSN 0142-9612. PMC 5944364. PMID 29704747. doi:10.1016/j.biomaterials.2018.04.031. Consultado el 4 de junio de 2024.
- ↑ Jain, Piyush K.; Lo, Justin H.; Rananaware, Santosh; Downing, Marco; Panda, Apekshya; Tai, Michelle; Raghavan, Srivatsan; Fleming, Heather E. et al. (14 de noviembre de 2019). «Non-viral delivery of CRISPR/Cas9 complex using CRISPR-GPS nanocomplexes». Nanoscale (en inglés) 11 (44): 21317-21323. ISSN 2040-3372. doi:10.1039/C9NR01786K. Consultado el 4 de junio de 2024.
- ↑ a b Yip, Bon Ham (2020-06). «Recent Advances in CRISPR/Cas9 Delivery Strategies». Biomolecules (en inglés) 10 (6): 839. ISSN 2218-273X. doi:10.3390/biom10060839. Consultado el 4 de junio de 2024.
- ↑ Bak, Rasmus O.; Porteus, Matthew H. (2017-07). «CRISPR-Mediated Integration of Large Gene Cassettes Using AAV Donor Vectors». Cell Reports 20 (3): 750-756. ISSN 2211-1247. doi:10.1016/j.celrep.2017.06.064. Consultado el 4 de junio de 2024.
- ↑ Schmidt, Florian; Grimm, Dirk (2015-02). «CRISPR genome engineering and viral gene delivery: A case of mutual attraction». Biotechnology Journal (en inglés) 10 (2): 258-272. ISSN 1860-6768. doi:10.1002/biot.201400529. Consultado el 4 de junio de 2024.
- ↑ Waxmonsky, Nicole. «CRISPR 101: Mammalian Expression Systems and Delivery Methods». blog.addgene.org (en inglés estadounidense). Consultado el 4 de junio de 2024.
- ↑ Mishra, Tarun; Bhardwaj, Vipin; Ahuja, Neha; Gadgil, Pallavi; Ramdas, Pavitra; Shukla, Sanjeev; Chande, Ajit (2022-06). «Improved loss-of-function CRISPR-Cas9 genome editing in human cells concomitant with inhibition of TGF-β signaling». Molecular Therapy - Nucleic Acids 28: 202-218. ISSN 2162-2531. PMC 8961078. PMID 35402072. doi:10.1016/j.omtn.2022.03.003. Consultado el 4 de junio de 2024.
- ↑ Adli, Mazhar (15 de mayo de 2018). «The CRISPR tool kit for genome editing and beyond». Nature Communications (en inglés) 9 (1): 1911. ISSN 2041-1723. doi:10.1038/s41467-018-04252-2. Consultado el 4 de junio de 2024.
- ↑ Svitashev, Sergei; Schwartz, Christine; Lenderts, Brian; Young, Joshua K.; Mark Cigan, A. (16 de noviembre de 2016). «Genome editing in maize directed by CRISPR–Cas9 ribonucleoprotein complexes». Nature Communications (en inglés) 7 (1): 13274. ISSN 2041-1723. doi:10.1038/ncomms13274. Consultado el 4 de junio de 2024.
- ↑ Liang, Zhen; Chen, Kunling; Li, Tingdong; Zhang, Yi; Wang, Yanpeng; Zhao, Qian; Liu, Jinxing; Zhang, Huawei et al. (18 de enero de 2017). «Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes». Nature Communications (en inglés) 8 (1): 14261. ISSN 2041-1723. PMC 5253684. PMID 28098143. doi:10.1038/ncomms14261. Consultado el 4 de junio de 2024.
- ↑ Cui, Zifeng; Tian, Rui; Huang, Zhaoyue; Jin, Zhuang; Li, Lifang; Liu, Jiashuo; Huang, Zheying; Xie, Hongxian et al. (17 de marzo de 2022). «FrCas9 is a CRISPR/Cas9 system with high editing efficiency and fidelity». Nature Communications (en inglés) 13 (1): 1425. ISSN 2041-1723. PMC 8931148. PMID 35301321. doi:10.1038/s41467-022-29089-8. Consultado el 4 de junio de 2024.
- ↑ Kleinstiver, Benjamin P.; Pattanayak, Vikram; Prew, Michelle S.; Tsai, Shengdar Q.; Nguyen, Nhu T.; Zheng, Zongli; Joung, J. Keith (2016-01). «High-fidelity CRISPR–Cas9 nucleases with no detectable genome-wide off-target effects». Nature (en inglés) 529 (7587): 490-495. ISSN 1476-4687. doi:10.1038/nature16526. Consultado el 4 de junio de 2024.
- ↑ Slaymaker, Ian M.; Gao, Linyi; Zetsche, Bernd; Scott, David A.; Yan, Winston X.; Zhang, Feng (2016-01). «Rationally engineered Cas9 nucleases with improved specificity». Science (en inglés) 351 (6268): 84-88. ISSN 0036-8075. doi:10.1126/science.aad5227. Consultado el 4 de junio de 2024.
- ↑ Yin, Hao; Song, Chun-Qing; Suresh, Sneha; Kwan, Suet-Yan; Wu, Qiongqiong; Walsh, Stephen; Ding, Junmei; Bogorad, Roman L. et al. (2018-03). «Partial DNA-guided Cas9 enables genome editing with reduced off-target activity». Nature Chemical Biology (en inglés) 14 (3): 311-316. ISSN 1552-4469. PMC 5902734. PMID 29377001. doi:10.1038/nchembio.2559. Consultado el 4 de junio de 2024.
- ↑ Riesenberg, Stephan; Helmbrecht, Nelly; Kanis, Philipp; Maricic, Tomislav; Pääbo, Svante (25 de enero de 2022). «Improved gRNA secondary structures allow editing of target sites resistant to CRISPR-Cas9 cleavage». Nature Communications (en inglés) 13 (1): 489. ISSN 2041-1723. PMC 8789806. PMID 35078986. doi:10.1038/s41467-022-28137-7. Consultado el 4 de junio de 2024.
- ↑ Fu, Yanfang; Sander, Jeffry D.; Reyon, Deepak; Cascio, Vincent M.; Joung, J. Keith (2014-03). «Improving CRISPR-Cas nuclease specificity using truncated guide RNAs». Nature Biotechnology (en inglés) 32 (3): 279-284. ISSN 1546-1696. PMC 3988262. PMID 24463574. doi:10.1038/nbt.2808. Consultado el 4 de junio de 2024.
- ↑ Thean, Dawn G. L.; Chu, Hoi Yee; Fong, John H. C.; Chan, Becky K. C.; Zhou, Peng; Kwok, Cynthia C. S.; Chan, Yee Man; Mak, Silvia Y. L. et al. (25 de abril de 2022). «Machine learning-coupled combinatorial mutagenesis enables resource-efficient engineering of CRISPR-Cas9 genome editor activities». Nature Communications (en inglés) 13 (1): 2219. ISSN 2041-1723. doi:10.1038/s41467-022-29874-5. Consultado el 4 de junio de 2024.
- ↑ Cho, Seung Woo; Kim, Sojung; Kim, Yongsub; Kweon, Jiyeon; Kim, Heon Seok; Bae, Sangsu; Kim, Jin-Soo (1 de enero de 2014). «Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases». Genome Research (en inglés) 24 (1): 132-141. ISSN 1088-9051. PMID 24253446. doi:10.1101/gr.162339.113. Consultado el 4 de junio de 2024.
- ↑ a b Oakes, Benjamin L.; Nadler, Dana C.; Flamholz, Avi; Fellmann, Christof; Staahl, Brett T.; Doudna, Jennifer A.; Savage, David F. (2016-06). «Profiling of engineering hotspots identifies an allosteric CRISPR-Cas9 switch». Nature Biotechnology (en inglés) 34 (6): 646-651. ISSN 1546-1696. PMC 4900928. PMID 27136077. doi:10.1038/nbt.3528. Consultado el 4 de junio de 2024.
- ↑ Nuñez, James K.; Harrington, Lucas B.; Doudna, Jennifer A. (18 de marzo de 2016). «Chemical and Biophysical Modulation of Cas9 for Tunable Genome Engineering». ACS Chemical Biology (en inglés) 11 (3): 681-688. ISSN 1554-8929. doi:10.1021/acschembio.5b01019. Consultado el 4 de junio de 2024.
- ↑ Zhou, Wenyuan; Deiters, Alexander (25 de abril de 2016). «Conditional Control of CRISPR/Cas9 Function». Angewandte Chemie International Edition (en inglés) 55 (18): 5394-5399. ISSN 1433-7851. doi:10.1002/anie.201511441. Consultado el 4 de junio de 2024.
- ↑ Polstein, Lauren R.; Gersbach, Charles A. (2015-03). «A light-inducible CRISPR-Cas9 system for control of endogenous gene activation». Nature Chemical Biology (en inglés) 11 (3): 198-200. ISSN 1552-4469. doi:10.1038/nchembio.1753. Consultado el 4 de junio de 2024.
- ↑ Nihongaki, Yuta; Yamamoto, Shun; Kawano, Fuun; Suzuki, Hideyuki; Sato, Moritoshi (19 de febrero de 2015). «CRISPR-Cas9-based Photoactivatable Transcription System». Chemistry & Biology 22 (2): 169-174. ISSN 1074-5521. doi:10.1016/j.chembiol.2014.12.011. Consultado el 4 de junio de 2024.
- ↑ Wright, Addison V.; Sternberg, Samuel H.; Taylor, David W.; Staahl, Brett T.; Bardales, Jorge A.; Kornfeld, Jack E.; Doudna, Jennifer A. (10 de marzo de 2015). «Rational design of a split-Cas9 enzyme complex». Proceedings of the National Academy of Sciences (en inglés) 112 (10): 2984-2989. ISSN 0027-8424. PMC 4364227. PMID 25713377. doi:10.1073/pnas.1501698112. Consultado el 4 de junio de 2024.
- ↑ Nihongaki, Yuta; Kawano, Fuun; Nakajima, Takahiro; Sato, Moritoshi (2015-07). «Photoactivatable CRISPR-Cas9 for optogenetic genome editing». Nature Biotechnology (en inglés) 33 (7): 755-760. ISSN 1546-1696. doi:10.1038/nbt.3245. Consultado el 4 de junio de 2024.
- ↑ Hemphill, James; Borchardt, Erin K.; Brown, Kalyn; Asokan, Aravind; Deiters, Alexander (6 de mayo de 2015). «Optical Control of CRISPR/Cas9 Gene Editing». Journal of the American Chemical Society (en inglés) 137 (17): 5642-5645. ISSN 0002-7863. doi:10.1021/ja512664v. Consultado el 4 de junio de 2024.
- ↑ Jain, Piyush K.; Ramanan, Vyas; Schepers, Arnout G.; Dalvie, Nisha S.; Panda, Apekshya; Fleming, Heather E.; Bhatia, Sangeeta N. (26 de septiembre de 2016). «Development of Light‐Activated CRISPR Using Guide RNAs with Photocleavable Protectors». Angewandte Chemie International Edition (en inglés) 55 (40): 12440-12444. ISSN 1433-7851. PMC 5864249. PMID 27554600. doi:10.1002/anie.201606123. Consultado el 4 de junio de 2024.
- ↑ Davis, Kevin M.; Pattanayak, Vikram; Thompson, David B.; Zuris, John A.; Liu, David R. (2015-05). «Small molecule–triggered Cas9 protein with improved genome-editing specificity». Nature Chemical Biology (en inglés) 11 (5): 316-318. ISSN 1552-4469. PMC 4402137. PMID 25848930. doi:10.1038/nchembio.1793. Consultado el 4 de junio de 2024.
- ↑ Liu, Kaiwen Ivy; Ramli, Muhammad Nadzim Bin; Woo, Cheok Wei Ariel; Wang, Yuanming; Zhao, Tianyun; Zhang, Xiujun; Yim, Guo Rong Daniel; Chong, Bao Yi et al. (2016-11). «A chemical-inducible CRISPR–Cas9 system for rapid control of genome editing». Nature Chemical Biology (en inglés) 12 (11): 980-987. ISSN 1552-4469. doi:10.1038/nchembio.2179. Consultado el 4 de junio de 2024.
- ↑ Truong, Dong-Jiunn Jeffery; Kühner, Karin; Kühn, Ralf; Werfel, Stanislas; Engelhardt, Stefan; Wurst, Wolfgang; Ortiz, Oskar (16 de junio de 2015). «Development of an intein-mediated split–Cas9 system for gene therapy». Nucleic Acids Research 43 (13): 6450-6458. ISSN 0305-1048. PMC 4513872. PMID 26082496. doi:10.1093/nar/gkv601. Consultado el 4 de junio de 2024.
- ↑ Zetsche, Bernd; Volz, Sara E.; Zhang, Feng (2015-02). «A split-Cas9 architecture for inducible genome editing and transcription modulation». Nature Biotechnology (en inglés) 33 (2): 139-142. ISSN 1546-1696. doi:10.1038/nbt.3149. Consultado el 4 de junio de 2024.
- ↑ González, Federico; Zhu, Zengrong; Shi, Zhong-Dong; Lelli, Katherine; Verma, Nipun; Li, Qing V.; Huangfu, Danwei (2014-08). «An iCRISPR Platform for Rapid, Multiplexable, and Inducible Genome Editing in Human Pluripotent Stem Cells». Cell Stem Cell 15 (2): 215-226. ISSN 1934-5909. doi:10.1016/j.stem.2014.05.018. Consultado el 4 de junio de 2024.
- ↑ Dow, Lukas E.; Fisher, Jonathan; O'Rourke, Kevin P.; Muley, Ashlesha; Kastenhuber, Edward R.; Livshits, Geulah; Tschaharganeh, Darjus F.; Socci, Nicholas D. et al. (2015-04). «Inducible in vivo genome editing with CRISPR-Cas9». Nature Biotechnology (en inglés) 33 (4): 390-394. ISSN 1546-1696. PMC 4390466. PMID 25690852. doi:10.1038/nbt.3155. Consultado el 4 de junio de 2024.
- ↑ Yu, Chen; Liu, Yanxia; Ma, Tianhua; Liu, Kai; Xu, Shaohua; Zhang, Yu; Liu, Honglei; La Russa, Marie et al. (2015-02). «Small Molecules Enhance CRISPR Genome Editing in Pluripotent Stem Cells». Cell Stem Cell 16 (2): 142-147. ISSN 1934-5909. doi:10.1016/j.stem.2015.01.003. Consultado el 4 de junio de 2024.
- ↑ Schimmel, Joost; Muñoz-Subirana, Núria; Kool, Hanneke; van Schendel, Robin; van der Vlies, Sven; Kamp, Juliette A.; de Vrij, Femke M.S.; Kushner, Steven A. et al. (2023-02). «Modulating mutational outcomes and improving precise gene editing at CRISPR-Cas9-induced breaks by chemical inhibition of end-joining pathways». Cell Reports 42 (2): 112019. ISSN 2211-1247. doi:10.1016/j.celrep.2023.112019. Consultado el 4 de junio de 2024.
- ↑ Maruyama, Takeshi; Dougan, Stephanie K.; Truttmann, Matthias C.; Bilate, Angelina M.; Ingram, Jessica R.; Ploegh, Hidde L. (2015-05). «Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining». Nature Biotechnology (en inglés) 33 (5): 538-542. ISSN 1546-1696. PMC 4618510. PMID 25798939. doi:10.1038/nbt.3190. Consultado el 4 de junio de 2024.
- ↑ a b Ma, Xiaojie; Chen, Xi; Jin, Yan; Ge, Wenyan; Wang, Weiyun; Kong, Linghao; Ji, Junfang; Guo, Xing et al. (3 de abril de 2018). «Small molecules promote CRISPR-Cpf1-mediated genome editing in human pluripotent stem cells». Nature Communications (en inglés) 9 (1): 1303. ISSN 2041-1723. PMC 5880812. PMID 29610531. doi:10.1038/s41467-018-03760-5. Consultado el 4 de junio de 2024.
- ↑ Nishida, Keiji; Arazoe, Takayuki; Yachie, Nozomu; Banno, Satomi; Kakimoto, Mika; Tabata, Mayura; Mochizuki, Masao; Miyabe, Aya et al. (16 de septiembre de 2016). «Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems». Science (en inglés) 353 (6305). ISSN 0036-8075. doi:10.1126/science.aaf8729. Consultado el 4 de junio de 2024.
- ↑ Komor, Alexis C.; Kim, Yongjoo B.; Packer, Michael S.; Zuris, John A.; Liu, David R. (2016-05). «Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage». Nature (en inglés) 533 (7603): 420-424. ISSN 1476-4687. PMC 4873371. PMID 27096365. doi:10.1038/nature17946. Consultado el 4 de junio de 2024.
- ↑ Gaudelli, Nicole M.; Komor, Alexis C.; Rees, Holly A.; Packer, Michael S.; Badran, Ahmed H.; Bryson, David I.; Liu, David R. (2017-11). «Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage». Nature (en inglés) 551 (7681): 464-471. ISSN 1476-4687. PMC 5726555. PMID 29160308. doi:10.1038/nature24644. Consultado el 4 de junio de 2024.
- ↑ Chen, Liwei; Park, Jung Eun; Paa, Peter; Rajakumar, Priscilla D.; Prekop, Hong-Ting; Chew, Yi Ting; Manivannan, Swathi N.; Chew, Wei Leong (2 de marzo de 2021). «Programmable C:G to G:C genome editing with CRISPR-Cas9-directed base excision repair proteins». Nature Communications (en inglés) 12 (1): 1384. ISSN 2041-1723. PMC 7925527. PMID 33654077. doi:10.1038/s41467-021-21559-9. Consultado el 4 de junio de 2024.
- ↑ a b c Kurata, Morito; Yamamoto, Kouhei; Moriarity, Branden S.; Kitagawa, Masanobu; Largaespada, David A. (2018-02). «CRISPR/Cas9 library screening for drug target discovery». Journal of Human Genetics (en inglés) 63 (2): 179-186. ISSN 1435-232X. doi:10.1038/s10038-017-0376-9. Consultado el 4 de junio de 2024.
- ↑ Gasiunas, Giedrius; Barrangou, Rodolphe; Horvath, Philippe; Siksnys, Virginijus (25 de septiembre de 2012). «Cas9–crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria». Proceedings of the National Academy of Sciences (en inglés) 109 (39). ISSN 0027-8424. doi:10.1073/pnas.1208507109. Consultado el 4 de junio de 2024.
- ↑ Satomura, Atsushi; Nishioka, Ryosuke; Mori, Hitoshi; Sato, Kosuke; Kuroda, Kouichi; Ueda, Mitsuyoshi (18 de mayo de 2017). «Precise genome-wide base editing by the CRISPR Nickase system in yeast». Scientific Reports (en inglés) 7 (1): 2095. ISSN 2045-2322. PMC 5437071. PMID 28522803. doi:10.1038/s41598-017-02013-7. Consultado el 4 de junio de 2024.
- ↑ Hiranniramol, Kasidet; Chen, Yuhao; Liu, Weijun; Wang, Xiaowei (23 de enero de 2020). «Generalizable sgRNA design for improved CRISPR/Cas9 editing efficiency». Bioinformatics 36 (9): 2684-2689. ISSN 1367-4803. PMC 7203743. PMID 31971562. doi:10.1093/bioinformatics/btaa041. Consultado el 4 de junio de 2024.
- ↑ Yu, Jason S.L.; Yusa, Kosuke (2019-07). «Genome-wide CRISPR-Cas9 screening in mammalian cells». Methods. 164-165: 29-35. ISSN 1046-2023. doi:10.1016/j.ymeth.2019.04.015. Consultado el 4 de junio de 2024.
- ↑ a b c Agrotis, Alexander; Ketteler, Robin (24 de septiembre de 2015). «A new age in functional genomics using CRISPR/Cas9 in arrayed library screening». Frontiers in Genetics (en english) 6. ISSN 1664-8021. doi:10.3389/fgene.2015.00300. Consultado el 4 de junio de 2024.
- ↑ Joung, Julia; Konermann, Silvana; Gootenberg, Jonathan S.; Abudayyeh, Omar O.; Platt, Randall J.; Brigham, Mark D.; Sanjana, Neville E.; Zhang, Feng (2017-04). «Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening». Nature Protocols (en inglés) 12 (4): 828-863. ISSN 1750-2799. PMC 5526071. PMID 28333914. doi:10.1038/nprot.2017.016. Consultado el 4 de junio de 2024.
- ↑ Joung, Julia; Konermann, Silvana; Gootenberg, Jonathan S.; Abudayyeh, Omar O.; Platt, Randall J.; Brigham, Mark D.; Sanjana, Neville E.; Zhang, Feng (2017-04). «Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening». Nature Protocols (en inglés) 12 (4): 828-863. ISSN 1750-2799. PMC 5526071. PMID 28333914. doi:10.1038/nprot.2017.016. Consultado el 4 de junio de 2024.
- ↑ «Addgene: Pooled Libraries». www.addgene.org (en inglés). Consultado el 5 de junio de 2024.
- ↑ McDade, Joel R.; Waxmonsky, Nicole C.; Swanson, Lianna E.; Fan, Melina (2016-07). «Practical Considerations for Using Pooled Lentiviral CRISPR Libraries». Current Protocols in Molecular Biology (en inglés) 115 (1). ISSN 1934-3639. doi:10.1002/cpmb.8. Consultado el 5 de junio de 2024.
- ↑ Cheng, Albert W.; Wang, Haoyi; Yang, Hui; Shi, Linyu; Katz, Yarden; Theunissen, Thorold W.; Rangarajan, Sudharshan; Shivalila, Chikdu S. et al. (2013-10). «Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system». Cell Research (en inglés) 23 (10): 1163-1171. ISSN 1748-7838. PMC 3790238. PMID 23979020. doi:10.1038/cr.2013.122. Consultado el 5 de junio de 2024.
- ↑ Gilbert, Luke A.; Horlbeck, Max A.; Adamson, Britt; Villalta, Jacqueline E.; Chen, Yuwen; Whitehead, Evan H.; Guimaraes, Carla; Panning, Barbara et al. (2014-10). «Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation». Cell 159 (3): 647-661. ISSN 0092-8674. PMC 4253859. PMID 25307932. doi:10.1016/j.cell.2014.09.029. Consultado el 5 de junio de 2024.
- ↑ a b Dow, Lukas E. (2015-10). «Modeling Disease In Vivo With CRISPR/Cas9». Trends in Molecular Medicine 21 (10): 609-621. ISSN 1471-4914. PMC 4592741. PMID 26432018. doi:10.1016/j.molmed.2015.07.006. Consultado el 5 de junio de 2024.
- ↑ Doudna J, J.; Mali, P. (2016). CRISPR-Cas: A Laboratory Manual (en inglés). Nueva York: Cold Spring Harbor. ISBN 978-1-62182-130-4. OCLC 922914104.
- ↑ Zuo, Erwei; Huo, Xiaona; Yao, Xuan; Hu, Xinde; Sun, Yidi; Yin, Jianhang; He, Bingbing; Wang, Xing et al. (24 de noviembre de 2017). «CRISPR/Cas9-mediated targeted chromosome elimination». CRISPR Used to Eliminate Targeted Chromosomes in New Study (en inglés), Genome Web. Genome Biology 18 (1): 224. ISSN 1474-760X. PMC 5701507. PMID 29178945. doi:10.1186/s13059-017-1354-4. Consultado el 5 de junio de 2024.
- ↑ Javed, Muhammad R.; Sadaf, Maria; Ahmed, Temoor; Jamil, Amna; Nawaz, Marium; Abbas, Hira; Ijaz, Anam (1 de diciembre de 2018). «CRISPR-Cas System: History and Prospects as a Genome Editing Tool in Microorganisms». Current Microbiology (en inglés) 75 (12): 1675-1683. ISSN 1432-0991. doi:10.1007/s00284-018-1547-4. Consultado el 5 de junio de 2024.
- ↑ DiCarlo, James E.; Norville, Julie E.; Mali, Prashant; Rios, Xavier; Aach, John; Church, George M. (4 de marzo de 2013). «Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems». Nucleic Acids Research 41 (7): 4336-4343. ISSN 1362-4962. PMC 3627607. PMID 23460208. doi:10.1093/nar/gkt135. Consultado el 5 de junio de 2024.
- ↑ Giersch, Rachael M.; Finnigan, Gregory C. (2017-12). «Yeast Still a Beast: Diverse Applications of CRISPR/Cas Editing Technology in S. cerevisiae». The Yale Journal of Biology and Medicine 90 (4): 643-651. ISSN 1551-4056. PMC 5733842. PMID 29259528. Consultado el 5 de junio de 2024.
- ↑ Dhamad, Ahmed E.; Lessner, Daniel J. (15 de octubre de 2020). «A CRISPRi-dCas9 System for Archaea and Its Use To Examine Gene Function during Nitrogen Fixation by Methanosarcina acetivorans». En Atomi, Haruyuki, ed. Applied and Environmental Microbiology (en inglés) 86 (21). ISSN 0099-2240. doi:10.1128/AEM.01402-20. Consultado el 5 de junio de 2024.
- ↑ Raschmanová, Hana; Weninger, Astrid; Glieder, Anton; Kovar, Karin; Vogl, Thomas (2018-05). «Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects». Biotechnology Advances 36 (3): 641-665. ISSN 0734-9750. doi:10.1016/j.biotechadv.2018.01.006. Consultado el 5 de junio de 2024.
- ↑ a b Ma, Dongyuan; Liu, Feng (1 de diciembre de 2015). «Genome Editing and Its Applications in Model Organisms». Genomics, Proteomics & Bioinformatics 13 (6): 336-344. ISSN 1672-0229. PMC 4747648. PMID 26762955. doi:10.1016/j.gpb.2015.12.001. Consultado el 5 de junio de 2024.
- ↑ Khurshid, Haris; Jan, Sohail Ahmad; Shinwari, Zabta Khan; Jamal, Muhammad; Shah, Sabir Hussain (2018). «An Era of CRISPR/ Cas9 Mediated Plant Genome Editing». Current Issues in Molecular Biology (en inglés): 47-54. ISSN 1467-3037. doi:10.21775/cimb.026.047. Consultado el 5 de junio de 2024.
- ↑ Simone, Brandon W.; Martínez-Gálvez, Gabriel; WareJoncas, Zachary; Ekker, Stephen C. (2018-11). «Fishing for understanding: Unlocking the zebrafish gene editor’s toolbox». Methods 150: 3-10. ISSN 1046-2023. PMC 6590056. PMID 30076892. doi:10.1016/j.ymeth.2018.07.012. Consultado el 5 de junio de 2024.
- ↑ Singh, Priti; Schimenti, John C.; Bolcun-Filas, Ewelina (2015-01). «A mouse geneticist's practical guide to CRISPR applications». Genetics 199 (1): 1-15. ISSN 1943-2631. PMC 4286675. PMID 25271304. doi:10.1534/genetics.114.169771. Consultado el 5 de junio de 2024.
- ↑ Soni, Dheeraj; Wang, Dong-Mei; Regmi, Sushil C.; Mittal, Manish; Vogel, Stephen M.; Schlüter, Dirk; Tiruppathi, Chinnaswamy (16 de mayo de 2018). «Deubiquitinase function of A20 maintains and repairs endothelial barrier after lung vascular injury». Cell Death Discovery (en inglés) 4 (1): 1-14. ISSN 2058-7716. PMC 5955943. PMID 29796309. doi:10.1038/s41420-018-0056-3. Consultado el 5 de junio de 2024.
- ↑ Birling, Marie-Christine; Herault, Yann; Pavlovic, Guillaume (1 de agosto de 2017). «Modeling human disease in rodents by CRISPR/Cas9 genome editing». Mammalian Genome (en inglés) 28 (7): 291-301. ISSN 1432-1777. PMC 5569124. PMID 28677007. doi:10.1007/s00335-017-9703-x. Consultado el 5 de junio de 2024.
- ↑ Gao, Xue; Tao, Yong; Lamas, Veronica; Huang, Mingqian; Yeh, Wei-Hsi; Pan, Bifeng; Hu, Yu-Juan; Hu, Johnny H. et al. (2018-01). «Treatment of autosomal dominant hearing loss by in vivo delivery of genome editing agents». Nature (en inglés) 553 (7687): 217-221. ISSN 1476-4687. PMC 5784267. PMID 29258297. doi:10.1038/nature25164. Consultado el 5 de junio de 2024.
- ↑ Kadam, Ulhas Sopanrao; Shelake, Rahul Mahadev; Chavhan, Rahul L.; Suprasanna, Penna (2018-10). «Concerns regarding ‘off-target’ activity of genome editing endonucleases». Plant Physiology and Biochemistry 131: 22-30. ISSN 0981-9428. doi:10.1016/j.plaphy.2018.03.027. Consultado el 5 de junio de 2024.
- ↑ Kimberland, Michelle L.; Hou, Wangfang; Alfonso-Pecchio, Adolfo; Wilson, Stephen; Rao, Yanhua; Zhang, Shu; Lu, Quinn (2018-10). «Strategies for controlling CRISPR/Cas9 off-target effects and biological variations in mammalian genome editing experiments». Journal of Biotechnology 284: 91-101. ISSN 0168-1656. doi:10.1016/j.jbiotec.2018.08.007. Consultado el 5 de junio de 2024.
- ↑ van Erp, Paul BG; Bloomer, Gary; Wilkinson, Royce; Wiedenheft, Blake (2015-06). «The history and market impact of CRISPR RNA-guided nucleases». Current Opinion in Virology 12: 85-90. ISSN 1879-6257. PMC 4470805. PMID 25914022. doi:10.1016/j.coviro.2015.03.011. Consultado el 5 de junio de 2024.
- ↑ Maggio, Ignazio; Gonçalves, Manuel A.F.V. (2015-05). «Genome editing at the crossroads of delivery, specificity, and fidelity». Trends in Biotechnology 33 (5): 280-291. ISSN 0167-7799. doi:10.1016/j.tibtech.2015.02.011. Consultado el 5 de junio de 2024.
- ↑ Rath, Devashish; Amlinger, Lina; Rath, Archana; Lundgren, Magnus (2015-10). «The CRISPR-Cas immune system: Biology, mechanisms and applications». Biochimie 117: 119-128. ISSN 0300-9084. doi:10.1016/j.biochi.2015.03.025. Consultado el 5 de junio de 2024.
- ↑ «What Is CRISPR? How Does It Work? Is It Gene Editing? » LiveScience.Tech». LiveScience.Tech (en inglés estadounidense). 30 de abril de 2018. Archivado desde el original el 6 de febrero de 2020. Consultado el 5 de junio de 2024.
- ↑ a b Freedman, Benjamin S.; Brooks, Craig R.; Lam, Albert Q.; Fu, Hongxia; Morizane, Ryuji; Agrawal, Vishesh; Saad, Abdelaziz F.; Li, Michelle K. et al. (23 de octubre de 2015). «Modelling kidney disease with CRISPR-mutant kidney organoids derived from human pluripotent epiblast spheroids». Nature Communications (en inglés) 6 (1): 8715. ISSN 2041-1723. PMC 4620584. PMID 26493500. doi:10.1038/ncomms9715. Consultado el 5 de junio de 2024.
- ↑ Cruz, Nelly M.; Song, Xuewen; Czerniecki, Stefan M.; Gulieva, Ramila E.; Churchill, Angela J.; Kim, Yong Kyun; Winston, Kosuke; Tran, Linh M. et al. (2017-11). «Organoid cystogenesis reveals a critical role of microenvironment in human polycystic kidney disease». Nature Materials (en inglés) 16 (11): 1112-1119. ISSN 1476-4660. PMC 5936694. PMID 28967916. doi:10.1038/nmat4994. Consultado el 5 de junio de 2024.
- ↑ Kim, Yong Kyun; Refaeli, Ido; Brooks, Craig R.; Jing, Peifeng; Gulieva, Ramila E.; Hughes, Michael R.; Cruz, Nelly M.; Liu, Yannan et al. (29 de septiembre de 2017). «Gene-Edited Human Kidney Organoids Reveal Mechanisms of Disease in Podocyte Development». Stem Cells 35 (12): 2366-2378. ISSN 1066-5099. PMC 5742857. PMID 28905451. doi:10.1002/stem.2707. Consultado el 5 de junio de 2024.
- ↑ Bellin, Milena; Casini, Simona; Davis, Richard P; D'Aniello, Cristina; Haas, Jessica; Ward-van Oostwaard, Dorien; Tertoolen, Leon G J; Jung, Christian B et al. (11 de diciembre de 2013). «Isogenic human pluripotent stem cell pairs reveal the role of a KCNH2 mutation in long-QT syndrome: Isogenic pairs of LQT2 pluripotent stem cells». The EMBO Journal (en inglés) 32 (24): 3161-3175. doi:10.1038/emboj.2013.240. Consultado el 5 de junio de 2024.
- ↑ Cai, Liquan; Fisher, Alfred L.; Huang, Haochu; Xie, Zijian (2016-12). «CRISPR-mediated genome editing and human diseases». Genes & Diseases 3 (4): 244-251. ISSN 2352-3042. doi:10.1016/j.gendis.2016.07.003. Consultado el 5 de junio de 2024.
- ↑ Fernández, Clara Rodríguez (4 de abril de 2024). «Seven diseases CRISPR technology could cure». Labiotech.eu (en inglés estadounidense). Consultado el 5 de junio de 2024.
- ↑ a b «CRISPR/Cas9 and Cancer». Immuno-Oncology News (en inglés estadounidense). Consultado el 5 de junio de 2024.
- ↑ Crossley, Merlin (1 de febrero de 2021). «New CRISPR technology could revolutionise gene therapy, offering new hope to people with genetic diseases». The Conversation (en inglés estadounidense). Consultado el 5 de junio de 2024.
- ↑ Cromer, M. Kyle; Camarena, Joab; Martin, Renata M.; Lesch, Benjamin J.; Vakulskas, Christopher A.; Bode, Nicole M.; Kurgan, Gavin; Collingwood, Michael A. et al. (2021-04). «Gene replacement of α-globin with β-globin restores hemoglobin balance in β-thalassemia-derived hematopoietic stem and progenitor cells». Nature Medicine (en inglés) 27 (4): 677-687. ISSN 1546-170X. PMC 8265212. PMID 33737751. doi:10.1038/s41591-021-01284-y. Consultado el 5 de junio de 2024.
- ↑ Xie, Fei; Ye, Lin; Chang, Judy C.; Beyer, Ashley I.; Wang, Jiaming; Muench, Marcus O.; Kan, Yuet Wai (1 de septiembre de 2014). «Seamless gene correction of β-thalassemia mutations in patient-specific iPSCs using CRISPR/Cas9 and piggyBac». Genome Research (en inglés) 24 (9): 1526-1533. ISSN 1088-9051. PMC 4158758. PMID 25096406. doi:10.1101/gr.173427.114. Consultado el 5 de junio de 2024.
- ↑ a b Frangoul, Haydar; Altshuler, David; Cappellini, M. Domenica; Chen, Yi-Shan; Domm, Jennifer; Eustace, Brenda K.; Foell, Juergen; de la Fuente, Josu et al. (21 de enero de 2021). «CRISPR-Cas9 Gene Editing for Sickle Cell Disease and β-Thalassemia». New England Journal of Medicine (en inglés) 384 (3): 252-260. ISSN 0028-4793. doi:10.1056/NEJMoa2031054. Consultado el 5 de junio de 2024.
- ↑ Dever, Daniel P.; Bak, Rasmus O.; Reinisch, Andreas; Camarena, Joab; Washington, Gabriel; Nicolas, Carmencita E.; Pavel-Dinu, Mara; Saxena, Nivi et al. (1 de noviembre de 2016). «CRISPR/Cas9 β-globin gene targeting in human haematopoietic stem cells». Nature 539: 384-389. ISSN 0028-0836. doi:10.1038/nature20134. Consultado el 5 de junio de 2024.
- ↑ «CRISPR “One Shot" Cell Therapy for Hemophilia Developed». GEN - Genetic Engineering and Biotechnology News (en inglés estadounidense). 2 de mayo de 2018. Consultado el 5 de junio de 2024.
- ↑ Marangi, Michele; Pistritto, Giuseppa (20 de abril de 2018). «Innovative Therapeutic Strategies for Cystic Fibrosis: Moving Forward to CRISPR Technique». Frontiers in Pharmacology (en english) 9. ISSN 1663-9812. PMC 5920621. PMID 29731717. doi:10.3389/fphar.2018.00396. Consultado el 5 de junio de 2024.
- ↑ Bengtsson, Niclas E.; Hall, John K.; Odom, Guy L.; Phelps, Michael P.; Andrus, Colin R.; Hawkins, R. David; Hauschka, Stephen D.; Chamberlain, Joel R. et al. (14 de febrero de 2017). «Muscle-specific CRISPR/Cas9 dystrophin gene editing ameliorates pathophysiology in a mouse model for Duchenne muscular dystrophy». Nature Communications (en inglés) 8 (1): 14454. ISSN 2041-1723. doi:10.1038/ncomms14454. Consultado el 5 de junio de 2024.
- ↑ Eisenstein, Michael (1 de mayo de 2018). «CRISPR takes on Huntington's disease». Nature 557: S42-S43. ISSN 0028-0836. doi:10.1038/d41586-018-05177-y. Consultado el 5 de junio de 2024.
- ↑ Dabrowska, Magdalena; Juzwa, Wojciech; Krzyzosiak, Wlodzimierz J.; Olejniczak, Marta (26 de febrero de 2018). «Precise Excision of the CAG Tract from the Huntingtin Gene by Cas9 Nickases». Frontiers in Neuroscience (en english) 12. ISSN 1662-453X. PMC 5834764. PMID 29535594. doi:10.3389/fnins.2018.00075. Consultado el 5 de junio de 2024.
- ↑ King, Anthony (7 de marzo de 2018). «A CRISPR edit for heart disease». Nature (en inglés) 555 (7695): S23-S25. doi:10.1038/d41586-018-02482-4. Consultado el 5 de junio de 2024.
- ↑ Scudellari, Megan (9 de julio de 2019). «Self-destructing mosquitoes and sterilized rodents: the promise of gene drives». Nature (en inglés) 571 (7764): 160-162. doi:10.1038/d41586-019-02087-5. Consultado el 5 de junio de 2024.
- ↑ Abrahimi, Parwiz; Chang, William G.; Kluger, Martin S.; Qyang, Yibing; Tellides, George; Saltzman, W. Mark; Pober, Jordan S. (3 de julio de 2015). «Efficient Gene Disruption in Cultured Primary Human Endothelial Cells by CRISPR/Cas9». Circulation Research (en inglés) 117 (2): 121-128. ISSN 0009-7330. PMC 4490936. PMID 25940550. doi:10.1161/CIRCRESAHA.117.306290. Consultado el 5 de junio de 2024.
- ↑ «A Year In, 1st Patient To Get Gene Editing For Sickle Cell Disease Is Thriving». NPR.org (en inglés).
- ↑ «CRISPR technology to cure sickle cell disease». ScienceDaily (en inglés). Consultado el 5 de junio de 2024.
- ↑ Uddin, Fathema; Rudin, Charles M.; Sen, Triparna (7 de agosto de 2020). «CRISPR Gene Therapy: Applications, Limitations, and Implications for the Future». Frontiers in Oncology (en english) 10. ISSN 2234-943X. PMC 7427626. PMID 32850447. doi:10.3389/fonc.2020.01387. Consultado el 5 de junio de 2024.
- ↑ a b c d «CRISPR Clinical Trials: A 2022 Update». Innovative Genomics Institute (IGI) (en inglés estadounidense). Consultado el 5 de junio de 2024.
- ↑ «Editas Hits Pause on LCA10 Program in Search of Partner». BioSpace (en inglés estadounidense). Consultado el 5 de junio de 2024.
- ↑ Jensen, Trine I.; Axelgaard, Esben; Bak, Rasmus O. (2019-06). «Therapeutic gene editing in haematological disorders with CRISPR /Cas9». British Journal of Haematology (en inglés) 185 (5): 821-835. ISSN 0007-1048. doi:10.1111/bjh.15851. Consultado el 6 de junio de 2024.
- ↑ Reardon, Sara (22 de junio de 2016). «First CRISPR clinical trial gets green light from US panel». Nature (en inglés). ISSN 1476-4687. doi:10.1038/nature.2016.20137. Consultado el 6 de junio de 2024.
- ↑ Rosenblum, Daniel; Gutkin, Anna; Kedmi, Ranit; Ramishetti, Srinivas; Veiga, Nuphar; Jacobi, Ashley M.; Schubert, Mollie S.; Friedmann-Morvinski, Dinorah et al. (20 de noviembre de 2020). «CRISPR-Cas9 genome editing using targeted lipid nanoparticles for cancer therapy». Science Advances (en inglés) 6 (47). ISSN 2375-2548. PMC 7673804. PMID 33208369. doi:10.1126/sciadv.abc9450. Consultado el 6 de junio de 2024.
- ↑ «Base editing: Revolutionary therapy clears girl's incurable cancer». BBC News (en inglés británico). 11 de diciembre de 2022. Consultado el 6 de junio de 2024.
- ↑ «'Designer cells' reverse one-year-old's cancer». BBC News (en inglés británico). 5 de noviembre de 2015. Consultado el 6 de junio de 2024.
- ↑ «B.C. researchers launching clinical trial for first genetically engineered stem cell-based therapy for type 1 diabetes». UBC News (en inglés estadounidense). 12 de mayo de 2022. Consultado el 6 de junio de 2024.
- ↑ «CRISPR Therapeutics and ViaCyte, Inc. Announce First Patient Dosed in…». CRISPR Therapeutics (en inglés). Consultado el 6 de junio de 2024.
- ↑ National Institute on Drug Abuse. «Antiretroviral Therapy Combined With CRISPR Gene Editing Can Eliminate HIV Infection in Mice | NIDA Archives». National Institute on Drug Abuse (en inglés). Consultado el 7 de junio de 2024.
- ↑ «First Clinical Trial of CRISPR-Based HIV Therapy Founded on Breakthrough Research at Lewis Katz School of Medicine | Lewis Katz School of Medicine at Temple University». medicine.temple.edu (en inglés). Consultado el 7 de junio de 2024.
- ↑ Excision BioTherapeutics (24 de marzo de 2022). «A Phase 1/2a, Sequential Cohort, Single Ascending Dose Study of the Safety, Tolerability, Biodistribution, and Pharmacodynamics of EBT 101 in Aviremic HIV-1 Infected Adults on Stable Antiretroviral Therapy» (en inglés).
- ↑ «Three people were gene-edited in an effort to cure their HIV. The result is unknown.». MIT Technology Review (en inglés). Consultado el 7 de junio de 2024.
- ↑ «HIV in cell culture can be completely eliminated using CRISPR-Cas gene editing technology, increasing hopes of cure». EurekAlert! (en inglés). Consultado el 7 de junio de 2024.
- ↑ «Scientists say they can cut HIV out of cells» (en inglés británico). 20 de marzo de 2024. Consultado el 7 de junio de 2024.
- ↑ Gomaa, Ahmed A.; Klumpe, Heidi E.; Luo, Michelle L.; Selle, Kurt; Barrangou, Rodolphe; Beisel, Chase L. (28 de febrero de 2014). «Programmable Removal of Bacterial Strains by Use of Genome-Targeting CRISPR-Cas Systems». En Lee, Sang Yup, ed. mBio (en inglés) 5 (1). ISSN 2161-2129. PMC 3903277. PMID 24473129. doi:10.1128/mBio.00928-13. Consultado el 7 de junio de 2024.
- ↑ Citorik, Robert J.; Mimee, Mark; Lu, Timothy K. (2014-11). «Sequence-specific antimicrobials using efficiently delivered RNA-guided nucleases». Nature Biotechnology (en inglés) 32 (11): 1141-1145. ISSN 1546-1696. PMC 4237163. PMID 25240928. doi:10.1038/nbt.3011. Consultado el 7 de junio de 2024.
- ↑ a b Gholizadeh, P.; Aghazadeh, M.; Asgharzadeh, M.; Kafil, H. S. (1 de noviembre de 2017). «Suppressing the CRISPR/Cas adaptive immune system in bacterial infections». European Journal of Clinical Microbiology & Infectious Diseases (en inglés) 36 (11): 2043-2051. ISSN 1435-4373. doi:10.1007/s10096-017-3036-2. Consultado el 7 de junio de 2024.
- ↑ Gibney, Elizabeth (1 de enero de 2018). «What to expect in 2018: science in the new year». Nature 553: 12-13. ISSN 0028-0836. doi:10.1038/d41586-018-00009-5. Consultado el 7 de junio de 2024.
- ↑ Taylor, P. (3 de enero de 2019). «J&J takes stake in Locus' CRISPR-based 'Pac-Man' antimicrobials». Fierce Biotech (en inglés).
- ↑ Reardon, Sara (1 de junio de 2017). «Modified viruses deliver death to antibiotic-resistant bacteria». Nature (en inglés) 546 (7660): 587-588. ISSN 1476-4687. doi:10.1038/nature.2017.22173. Consultado el 7 de junio de 2024.
- ↑ Diemen, Ferdy R. van; Kruse, Elisabeth M.; Hooykaas, Marjolein J. G.; Bruggeling, Carlijn E.; Schürch, Anita C.; Ham, Petra M. van; Imhof, Saskia M.; Nijhuis, Monique et al. (30 de junio de 2016). «CRISPR/Cas9-Mediated Genome Editing of Herpesviruses Limits Productive and Latent Infections». PLOS Pathogens (en inglés) 12 (6): e1005701. ISSN 1553-7374. PMC 4928872. PMID 27362483. doi:10.1371/journal.ppat.1005701. Consultado el 7 de junio de 2024.
- ↑ PLOS Media (4 de agosto de 2016), Using CRISPR to combat viral infections: a new way to treat herpes? (en inglés), consultado el 7 de junio de 2024.
- ↑ a b c Science News Staff (17 de diciembre de 2015). «And Science's Breakthrough of the Year is ...». news.sciencemag.org (en inglés).
- ↑ Mullin, E. «CRISPR Opens Up New Possibilities for Transplants Using Pig Organs». MIT Technology Review (en inglés). Consultado el 7 de junio de 2024.
- ↑ Feijo, Sara. «How Brown neuroscientists are using CRISPR to accelerate brain research — and more». Brown University (en inglés). Consultado el 7 de junio de 2024.
- ↑ a b De Plano, Laura M.; Calabrese, Giovanna; Conoci, Sabrina; Guglielmino, Salvatore P. P.; Oddo, Salvatore; Caccamo, Antonella (2022-01). «Applications of CRISPR-Cas9 in Alzheimer’s Disease and Related Disorders». International Journal of Molecular Sciences (en inglés) 23 (15): 8714. ISSN 1422-0067. PMC 9368966. PMID 35955847. doi:10.3390/ijms23158714. Consultado el 7 de junio de 2024.
- ↑ Erkut, Esra; Yokota, Toshifumi (2022-01). «CRISPR Therapeutics for Duchenne Muscular Dystrophy». International Journal of Molecular Sciences (en inglés) 23 (3): 1832. ISSN 1422-0067. PMC 8836469. PMID 35163754. doi:10.3390/ijms23031832. Consultado el 7 de junio de 2024.
- ↑ Yamagata, Tetsushi; Raveau, Matthieu; Kobayashi, Kenta; Miyamoto, Hiroyuki; Tatsukawa, Tetsuya; Ogiwara, Ikuo; Itohara, Shigeyoshi; Hensch, Takao K. et al. (1 de julio de 2020). «CRISPR/dCas9-based Scn1a gene activation in inhibitory neurons ameliorates epileptic and behavioral phenotypes of Dravet syndrome model mice». Neurobiology of Disease 141: 104954. ISSN 0969-9961. doi:10.1016/j.nbd.2020.104954. Consultado el 7 de junio de 2024.
- ↑ Pinson, Anneline; Xing, Lei; Namba, Takashi; Kalebic, Nereo; Peters, Jula; Oegema, Christina Eugster; Traikov, Sofia; Reppe, Katrin et al. (9 de septiembre de 2022). «Human TKTL1 implies greater neurogenesis in frontal neocortex of modern humans than Neanderthals». Science (en inglés) 377 (6611). ISSN 0036-8075. doi:10.1126/science.abl6422. Consultado el 7 de junio de 2024.
- ↑ Mora-Bermúdez, Felipe; Kanis, Philipp; Macak, Dominik; Peters, Jula; Naumann, Ronald; Xing, Lei; Sarov, Mihail; Winkler, Sylke et al. (29 de julio de 2022). «Longer metaphase and fewer chromosome segregation errors in modern human than Neanderthal brain development». Science Advances (en inglés) 8 (30). ISSN 2375-2548. PMC 9337762. PMID 35905187. doi:10.1126/sciadv.abn7702. Consultado el 7 de junio de 2024.
- ↑ «Scientists discover how humans develop larger brains than other apes». phys.org (en inglés). Archivado desde el original el 19 de abril de 2021. Consultado el 7 de junio de 2024.
- ↑ Benito-Kwiecinski, Silvia; Giandomenico, Stefano L.; Sutcliffe, Magdalena; Riis, Erlend S.; Freire-Pritchett, Paula; Kelava, Iva; Wunderlich, Stephanie; Martin, Ulrich et al. (2021-04). «An early cell shape transition drives evolutionary expansion of the human forebrain». Cell 184 (8): 2084-2102.e19. ISSN 0092-8674. PMC 8054913. PMID 33765444. doi:10.1016/j.cell.2021.02.050. Archivado desde el original el 16 de octubre de 2017. Consultado el 7 de junio de 2024.
- ↑ Sawal, I. «Mini brains genetically altered with CRISPR to be Neanderthal-like». New Scientist (en inglés estadounidense). Archivado desde el original el 10 de marzo de 2021. Consultado el 7 de junio de 2024.
- ↑ Trujillo, Cleber A.; Rice, Edward S.; Schaefer, Nathan K.; Chaim, Isaac A.; Wheeler, Emily C.; Madrigal, Assael A.; Buchanan, Justin; Preissl, Sebastian et al. (12 de febrero de 2021). «Reintroduction of the archaic variant of NOVA1 in cortical organoids alters neurodevelopment». Science (en inglés) 371 (6530). ISSN 0036-8075. PMC 8006534. PMID 33574182. doi:10.1126/science.aax2537. Consultado el 7 de junio de 2024.
- ↑ Maricic, Tomislav; Helmbrecht, Nelly; Riesenberg, Stephan; Macak, Dominik; Kanis, Philipp; Lackner, Martin; Pugach-Matveeva, Agrafena Daria; Pääbo, Svante (15 de octubre de 2021). «Comment on “Reintroduction of the archaic variant of NOVA1 in cortical organoids alters neurodevelopment”». Science (en inglés) 374 (6565). ISSN 0036-8075. doi:10.1126/science.abi6060. Consultado el 7 de junio de 2024.
- ↑ Herai, Roberto H.; Szeto, Ryan A.; Trujillo, Cleber A.; Muotri, Alysson R. (15 de octubre de 2021). «Response to Comment on “Reintroduction of the archaic variant of NOVA1 in cortical organoids alters neurodevelopment”». Science (en inglés) 374 (6565). ISSN 0036-8075. doi:10.1126/science.abi9881. Consultado el 7 de junio de 2024.
- ↑ Dominguez, Antonia A.; Lim, Wendell A.; Qi, Lei S. (2016-01). «Beyond editing: repurposing CRISPR–Cas9 for precision genome regulation and interrogation». Nature Reviews Molecular Cell Biology (en inglés) 17 (1): 5-15. ISSN 1471-0080. PMC 4922510. PMID 26670017. doi:10.1038/nrm.2015.2. Consultado el 7 de junio de 2024.
- ↑ Shalem, Ophir; Sanjana, Neville E.; Hartenian, Ella; Shi, Xi; Scott, David A.; Mikkelsen, Tarjei S.; Heckl, Dirk; Ebert, Benjamin L. et al. (3 de enero de 2014). «Genome-Scale CRISPR-Cas9 Knockout Screening in Human Cells». Science (en inglés) 343 (6166): 84-87. ISSN 0036-8075. PMC 4089965. PMID 24336571. doi:10.1126/science.1247005. Consultado el 7 de junio de 2024.
- ↑ a b Pennisi, Elizabeth (23 de agosto de 2013). «The CRISPR Craze». Science (en inglés) 341 (6148): 833-836. ISSN 0036-8075. doi:10.1126/science.341.6148.833. Consultado el 7 de junio de 2024.
- ↑ a b Zimmer C (3 de junio de 2016). «Scientists Find Form of CRISPR Gene Editing With New Capabilities». The New York Times (en inglés). ISSN 0362-4331.
- ↑ Pickar-Oliver, Adrian; Gersbach, Charles A. (2019-08). «The next generation of CRISPR–Cas technologies and applications». Nature Reviews Molecular Cell Biology (en inglés) 20 (8): 490-507. ISSN 1471-0080. PMC 7079207. PMID 31147612. doi:10.1038/s41580-019-0131-5. Consultado el 7 de junio de 2024.
- ↑ Basak, Jolly; Nithin, Chandran (19 de noviembre de 2015). «Targeting Non-Coding RNAs in Plants with the CRISPR-Cas Technology is a Challenge yet Worth Accepting». Frontiers in Plant Science (en english) 6. ISSN 1664-462X. PMC 4652605. PMID 26635829. doi:10.3389/fpls.2015.01001. Consultado el 7 de junio de 2024.
- ↑ a b Woolf, T M; Chase, J M; Stinchcomb, D T (29 de agosto de 1995). «Toward the therapeutic editing of mutated RNA sequences.». Proceedings of the National Academy of Sciences (en inglés) 92 (18): 8298-8302. ISSN 0027-8424. PMC 41144. PMID 7545300. doi:10.1073/pnas.92.18.8298. Consultado el 7 de junio de 2024.
- ↑ Montiel-Gonzalez, Maria Fernanda; Vallecillo-Viejo, Isabel; Yudowski, Guillermo A.; Rosenthal, Joshua J. C. (5 de noviembre de 2013). «Correction of mutations within the cystic fibrosis transmembrane conductance regulator by site-directed RNA editing». Proceedings of the National Academy of Sciences (en inglés) 110 (45): 18285-18290. ISSN 0027-8424. PMC 3831439. PMID 24108353. doi:10.1073/pnas.1306243110. Consultado el 7 de junio de 2024.
- ↑ Cox, David B. T.; Gootenberg, Jonathan S.; Abudayyeh, Omar O.; Franklin, Brian; Kellner, Max J.; Joung, Julia; Zhang, Feng (24 de noviembre de 2017). «RNA editing with CRISPR-Cas13». Science (en inglés) 358 (6366): 1019-1027. ISSN 0036-8075. PMC 5793859. PMID 29070703. doi:10.1126/science.aaq0180. Consultado el 7 de junio de 2024.
- ↑ Williams, Sarah. «Neuroscientists expand CRISPR toolkit with new, compact Cas7-11 enzyme». Massachusetts Institute of Technology (en inglés). Consultado el 7 de junio de 2024.
- ↑ Kato, Kazuki; Zhou, Wenyuan; Okazaki, Sae; Isayama, Yukari; Nishizawa, Tomohiro; Gootenberg, Jonathan S.; Abudayyeh, Omar O.; Nishimasu, Hiroshi (2022-06). «Structure and engineering of the type III-E CRISPR-Cas7-11 effector complex». Cell 185 (13): 2324-2337.e16. ISSN 0092-8674. doi:10.1016/j.cell.2022.05.003. Consultado el 7 de junio de 2024.
- ↑ Özcan, Ahsen; Krajeski, Rohan; Ioannidi, Eleonora; Lee, Brennan; Gardner, Apolonia; Makarova, Kira S.; Koonin, Eugene V.; Abudayyeh, Omar O. et al. (2021-09). «Programmable RNA targeting with the single-protein CRISPR effector Cas7-11». Nature (en inglés) 597 (7878): 720-725. ISSN 1476-4687. doi:10.1038/s41586-021-03886-5. Consultado el 7 de junio de 2024.
- ↑ Cross, R. (25 de marzo de 2019). «Watch out, CRISPR. The RNA editing race is on». Chemical & Engineering News (en inglés) 97 (12).
- ↑ Akbari, Omar S.; Bellen, Hugo J.; Bier, Ethan; Bullock, Simon L.; Burt, Austin; Church, George M.; Cook, Kevin R.; Duchek, Peter et al. (28 de agosto de 2015). «Safeguarding gene drive experiments in the laboratory». Science (en inglés) 349 (6251): 927-929. ISSN 0036-8075. PMC 4692367. PMID 26229113. doi:10.1126/science.aac7932. Consultado el 7 de junio de 2024.
- ↑ Caplan, Arthur L; Parent, Brendan; Shen, Michael; Plunkett, Carolyn (2015-11). «No time to waste—the ethical challenges created by CRISPR: CRISPR /Cas, being an efficient, simple, and cheap technology to edit the genome of any organism, raises many ethical and regulatory issues beyond the use to manipulate human germ line cells». EMBO reports (en inglés) 16 (11): 1421-1426. ISSN 1469-221X. PMC 4641494. PMID 26450575. doi:10.15252/embr.201541337. Consultado el 7 de junio de 2024.
- ↑ Oye, Kenneth A.; Esvelt, Kevin; Appleton, Evan; Catteruccia, Flaminia; Church, George; Kuiken, Todd; Lightfoot, Shlomiya Bar-Yam; McNamara, Julie et al. (1 de agosto de 2014). «Regulating gene drives». Science 345: 626-628. ISSN 0036-8075. doi:10.1126/science.1254287. Consultado el 7 de junio de 2024.
- ↑ Gu, W.; Crawford, E. D.; O’Donovan, B. D.; Wilson, M. R.; Chow, E. D.; Retallack, H.; DeRisi, J. L. (4 de marzo de 2016). «Depletion of Abundant Sequences by Hybridization (DASH): using Cas9 to remove unwanted high-abundance species in sequencing libraries and molecular counting applications». Genome Biology 17 (1): 41. ISSN 1474-760X. PMC 4778327. PMID 26944702. doi:10.1186/s13059-016-0904-5. Consultado el 7 de junio de 2024.
- ↑ a b Kungulovski, Goran; Jeltsch, Albert (1 de febrero de 2016). «Epigenome Editing: State of the Art, Concepts, and Perspectives». Trends in Genetics (en inglés) 32 (2): 101-113. ISSN 0168-9525. PMID 26732754. doi:10.1016/j.tig.2015.12.001. Consultado el 30 de abril de 2021.
- ↑ Laufer, Benjamin I.; Singh, Shiva M. (17 de septiembre de 2015). «Strategies for precision modulation of gene expression by epigenome editing: an overview». Epigenetics & Chromatin 8 (1): 34. ISSN 1756-8935. PMC 4574080. PMID 26388942. doi:10.1186/s13072-015-0023-7.
- ↑ «Unique CRISPR gene therapy offers opioid-free chronic pain treatment». New Atlas. 11 de marzo de 2021. Consultado el 18 de abril de 2021.
- ↑ Moreno, Ana M.; Alemán, Fernando; Catroli, Glaucilene F.; Hunt, Matthew; Hu, Michael; Dailamy, Amir; Pla, Andrew; Woller, Sarah A. et al. (10 de marzo de 2021). «Long-lasting analgesia via targeted in situ repression of NaV1.7 in mice». Science Translational Medicine (en inglés) 13 (584): eaay9056. ISSN 1946-6234. PMC 8830379. PMID 33692134. doi:10.1126/scitranslmed.aay9056.
- ↑ KAISER, JOCELYN (1 de junio de 2022). «Better than CRISPR? Another way to fix gene problems may be safer and more versatile». www.science.org (en inglés). Consultado el 21 de agosto de 2022.
- ↑ McDonnell, Sarah. «New CRISPR-based tool inserts large DNA sequences at desired sites in cells». Massachusetts Institute of Technology via phys.org (en inglés). Consultado el 7 de junio de 2024.
- ↑ Yarnall, Matthew T. N.; Ioannidi, Eleonora I.; Schmitt-Ulms, Cian; Krajeski, Rohan N.; Lim, Justin; Villiger, Lukas; Zhou, Wenyuan; Jiang, Kaiyi et al. (2023-04). «Drag-and-drop genome insertion of large sequences without double-strand DNA cleavage using CRISPR-directed integrases». Nature Biotechnology (en inglés) 41 (4): 500-512. ISSN 1546-1696. PMC 10257351. PMID 36424489. doi:10.1038/s41587-022-01527-4. Consultado el 7 de junio de 2024.
- ↑ Anzalone, Andrew V.; Randolph, Peyton B.; Davis, Jessie R.; Sousa, Alexander A.; Koblan, Luke W.; Levy, Jonathan M.; Chen, Peter J.; Wilson, Christopher et al. (2019-12). «Search-and-replace genome editing without double-strand breaks or donor DNA». Nature (en inglés) 576 (7785): 149-157. ISSN 1476-4687. PMC 6907074. PMID 31634902. doi:10.1038/s41586-019-1711-4. Consultado el 7 de junio de 2024.
- ↑ Magazine, Smithsonian. «A New Gene Editing Tool Could Make CRISPR More Precise». Smithsonian Magazine (en inglés). Consultado el 7 de junio de 2024.
- ↑ a b Cohen, J. «New 'prime' genome editor could surpass CRISPR.» (en inglés). Science Magazine.
- ↑ a b «New “Prime Editing” Method Makes Only Single-Stranded DNA Cuts». The Scientist Magazine® (en inglés). Consultado el 7 de junio de 2024.
- ↑ a b Gallagher, J. (21 de octubre de 2019). «Prime editing: DNA tool could correct 89% of genetic defects». BBC News (en inglés británico). Consultado el 7 de junio de 2024.
- ↑ Regalado, A. (5 de marzo de 2015). «Engineering the Perfect Baby». MIT Technology Review (en inglés). Archivado desde el original el 13 de marzo de 2015.
- ↑ Baltimore, David; Berg, Paul; Botchan, Michael; Carroll, Dana; Charo, R. Alta; Church, George; Corn, Jacob E.; Daley, George Q. et al. (3 de abril de 2015). «A prudent path forward for genomic engineering and germline gene modification». Science (en inglés) 348 (6230): 36-38. ISSN 0036-8075. PMC 4394183. PMID 25791083. doi:10.1126/science.aab1028. Consultado el 7 de junio de 2024.
- ↑ Lanphier, Edward; Urnov, Fyodor; Haecker, Sarah Ehlen; Werner, Michael; Smolenski, Joanna (2015-03). «Don’t edit the human germ line». Nature (en inglés) 519 (7544): 410-411. ISSN 1476-4687. doi:10.1038/519410a. Consultado el 7 de junio de 2024.
- ↑ Wade, Nicholas (19 de marzo de 2015). «Scientists Seek Ban on Method of Editing the Human Genome». The New York Times (en inglés estadounidense). ISSN 0362-4331. Consultado el 7 de junio de 2024. «The biologists writing in Science support continuing laboratory research with the technique, and few if any scientists believe it is ready for clinical use. / Los biólogos que escriben en Science apoyan la continuación de la investigación en laboratorio con esta técnica, y pocos científicos, si es que hay alguno, creen que está lista para su uso clínico.»
- ↑ Liang, Puping; Xu, Yanwen; Zhang, Xiya; Ding, Chenhui; Huang, Rui; Zhang, Zhen; Lv, Jie; Xie, Xiaowei et al. (18 de abril de 2015). «CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes». Protein & Cell 6 (5): 363-372. ISSN 1674-800X. PMC 4417674. PMID 25894090. doi:10.1007/s13238-015-0153-5. Consultado el 7 de junio de 2024.
- ↑ Kolata, G. (23 de abril de 2015). «Chinese Scientists Edit Genes of Human Embryos, Raising Concerns». New York Times (en inglés).
- ↑ a b Cyranoski, David; Reardon, Sara (22 de abril de 2015). «Chinese scientists genetically modify human embryos». Nature (en inglés). ISSN 1476-4687. doi:10.1038/nature.2015.17378. Consultado el 7 de junio de 2024.
- ↑ «Chinese Researchers Experiment with Making HIV-Proof Embryos». MIT Technology Review (en inglés). Consultado el 7 de junio de 2024.
- ↑ «International Summit on Gene Editing». National Academies of Sciences, Engineering, and Medicine (en inglés). 3 de diciembre de 2015. Consultado el 7 de junio de 2024.
- ↑ Brokowski, Carolyn (2018-04). «Do CRISPR Germline Ethics Statements Cut It?». The CRISPR Journal (en inglés) 1 (2): 115-125. ISSN 2573-1599. PMC 6694771. PMID 31021208. doi:10.1089/crispr.2017.0024. Consultado el 7 de junio de 2024.
- ↑ Begley, Sharon (28 de noviembre de 2018). «Amid uproar, Chinese scientist defends creating gene-edited babies». STAT (en inglés estadounidense). Consultado el 7 de junio de 2024.
- ↑ Sample, Ian (13 de marzo de 2019). «Scientists call for global moratorium on gene editing of embryos». The Guardian (en inglés británico). ISSN 0261-3077. Consultado el 7 de junio de 2024.
- ↑ Callaway, Ewen (1 de febrero de 2016). «UK scientists gain licence to edit genes in human embryos». Nature (en inglés) 530 (7588): 18-18. ISSN 1476-4687. doi:10.1038/nature.2016.19270. Consultado el 7 de junio de 2024.
- ↑ McHughen, Alan; Smyth, Stuart (2008-01). «US regulatory system for genetically modified [genetically modified organism (GMO), rDNA or transgenic] crop cultivars». Plant Biotechnology Journal (en inglés) 6 (1): 2-12. ISSN 1467-7644. doi:10.1111/j.1467-7652.2007.00300.x. Consultado el 7 de junio de 2024.
- ↑ USDA. «Re: Request to confirm» (en inglés).
- ↑ Waltz, Emily (1 de abril de 2016). «Gene-edited CRISPR mushroom escapes US regulation». Nature (en inglés) 532 (7599): 293-293. ISSN 1476-4687. doi:10.1038/nature.2016.19754. Consultado el 7 de junio de 2024.
- ↑ Ledford, Heidi (1 de abril de 2016). «Gene-editing surges as US rethinks regulations». Nature (en inglés) 532 (7598): 158-159. ISSN 1476-4687. doi:10.1038/532158a. Consultado el 7 de junio de 2024.
- ↑ Brown, Kristen V. (1 de febrero de 2017). «The FDA Is Cracking Down On Rogue Genetic Engineers». Gizmodo Australia (en inglés australiano). Consultado el 7 de junio de 2024.
- ↑ «Guidance for Industry #187 / Regulation of Intentionally Altered Genomic DNA in Animals». Food and Drug Administration (en inglés). 11 de febrero de 2020.
- ↑ Cyranoski, David (1 de agosto de 2017). «China’s embrace of embryo selection raises thorny questions». Nature (en inglés) 548 (7667): 272-274. ISSN 1476-4687. doi:10.1038/548272a. Consultado el 7 de junio de 2024.
- ↑ Peng, Yaojin (2016-06). «The morality and ethics governing CRISPR–Cas9 patents in China». Nature Biotechnology (en inglés) 34 (6): 616-618. ISSN 1546-1696. doi:10.1038/nbt.3590. Consultado el 7 de junio de 2024.
- ↑ Fan, Preetika Rana, Amy Dockser Marcus and Wenxin. «China, Unhampered by Rules, Races Ahead in Gene-Editing Trials». WSJ (en inglés estadounidense). Consultado el 7 de junio de 2024.
- ↑ «Precise Gene Editing in Plants/ 10 Breakthrough Technologies 2016». MIT Technology review (en inglés) (Massachusetts Institute of Technology). 2016. Archivado desde el original el 5 de noviembre de 2017.
- ↑ Larson, C, Schaffer, A (2014). «Genome Editing/ 10 Breakthrough Technologies 2014». Massachusetts Institute of Technology (en inglés). Archivado desde el original el 5 de diciembre de 2016.
- ↑ «Tang Prize | Laureates | Emmanuelle Charpentier». www.tang-prize.org (en inglés). Consultado el 7 de junio de 2024.
- ↑ «The Nobel Prize in Chemistry 2020». NobelPrize.org (en inglés estadounidense). Consultado el 7 de junio de 2024.